前言
最近总看到“分享脚本raw2pos.py”的公众号文章,这些文章标题相同,内容相同,就是贴了不知道谁人写的将 deepmd/raw 格式(dpgen 默认输出的结构格式)转化为 POSCAR 或者 xyz 的脚本。这类公众号甚至直接将代码作为文本粘贴,看得本人很恼火。
因为这些事情借助 dpdata,明明就是三行代码,甚至一行终端命令的事情。尤其想起前段时间跟曾晋哲老师聊天,想到他主持的很多很有意义的事情往往不被了解或是又被一堆人重造轮子,不觉唏嘘。
这篇文章就简单讲解一下如何用 dpdata 快速进行格式转化。同时也希望一定程度扭转一下整个行业对“祖传脚本”的依赖风气。
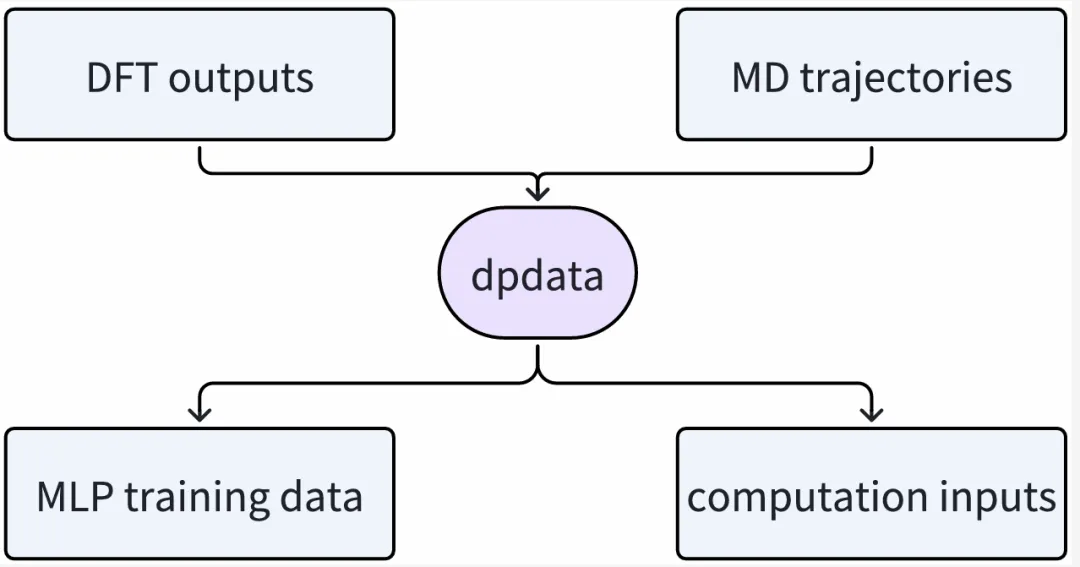 dpdata可以处理的数据类型
dpdata可以处理的数据类型关于dpdata
dpdata 项目于 2019 年启动,旨在提供用于表示与处理原子级机器学习数据集的工具,支持多种数据处理操作,并可在不同软件的 DFT 输出、分子动力学(MD)轨迹、机器学习势训练数据与计算输入之间进行格式互转。
随着项目推进,dpdata 引入插件机制,可灵活支持任意自定义格式的转换。
论文“dpdata: A Scalable Python Toolkit for Atomistic Machine Learning Data Sets”系统阐述了 dpdata 的功能与技术架构,展示了其下游应用,并对比了 dpdata 与 ASE 的性能;结果显示,得益于架构设计,dpdata 在内存占用与数据解析速度方面均具有显著优势。
dpdata 在 GitHub 开源,目前 dpdata 已更新到 v1.0.0 版本,进入成熟阶段。
dpdata 的文档:(可点击“阅读原文”跳转) https://docs.deepmodeling.com/projects/dpdata/en/master/
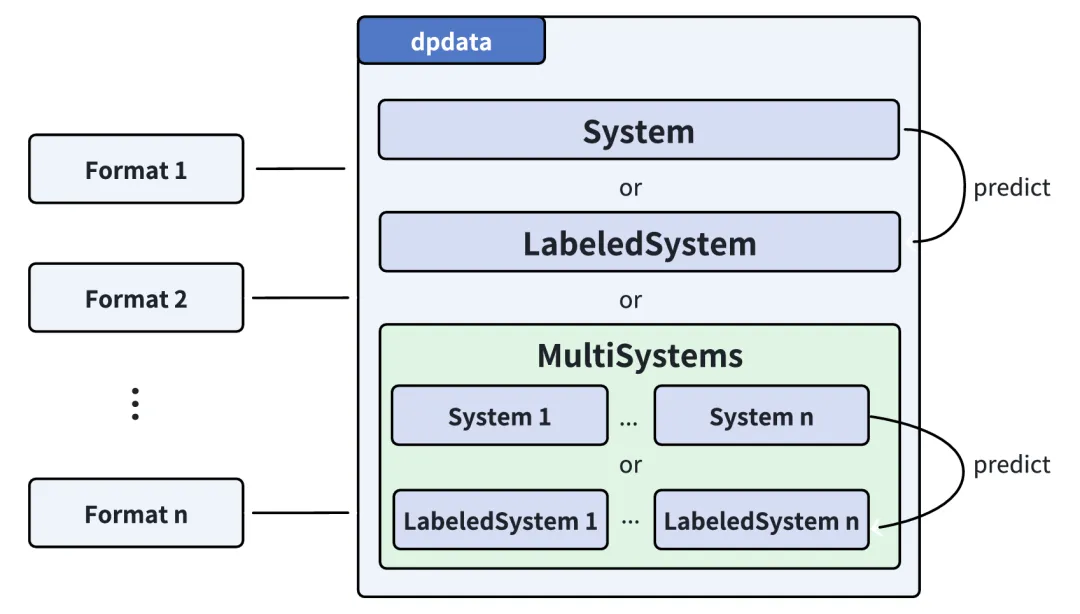 dpdata的系统对象设计
dpdata的系统对象设计安装dpdata
pip install dpdata
或者
conda install -c conda-forge dpdata
快速完成结构格式转换
案例准备
我们先用 ase 创建一个 POSCAR 格式的 Cu 晶体,用来做格式转换。如果没装 ase ,通过pip install即可。
from ase.io import write
from ase.build import bulk
Cu_bulk = bulk('Cu', 'fcc', a=3.61)
write('Cu_bulk.vasp', Cu_bulk)
这个POSCAR长相如下
Cu
1.0000000000000000
0.0000000000000000 1.8049999999999999 1.8049999999999999
1.8049999999999999 0.0000000000000000 1.8049999999999999
1.8049999999999999 1.8049999999999999 0.0000000000000000
Cu
1
Cartesian
0.0000000000000000 0.0000000000000000 0.0000000000000000
POSCAR -> deepmd
我们通过dpdata,将这个POSCAR转化为deepmd/raw和deepmd/npy格式
import dpdata
Cu_dpdata = dpdata.System("Cu_bulk.vasp", fmt="vasp/poscar")
Cu_dpdata.to(fmt="deepmd/raw", file_name="Cu_bulk_deepmd")
Cu_dpdata.to(fmt="deepmd/npy", file_name="Cu_bulk_deepmd")
上述代码的另一种写法:
import dpdata
Cu_dpdata = dpdata.System("Cu_bulk.vasp", fmt="vasp/poscar")
Cu_dpdata.to_deepmd_raw(file_name="Cu_bulk_deepmd")
Cu_dpdata.to_deepmd_npy(file_name="Cu_bulk_deepmd")
这样得到的文件如下:
Cu_bulk_deepmd> tree
.
├── box.raw
├── coord.raw
├── set.000
│ ├── box.npy
│ └── coord.npy
├── type.raw
└── type_map.raw
1 directory, 6 files
其中*.raw文件即为deepmd/raw文本文件格式的结构,set.000/*.npy文件即为deepmd/npy二进制格式的结构。这几个结构文件格式主要服务于 deepmd-kit 训练和 dpgen 主动学习。
其中.npy的二进制格式压缩度非常高,如果采用deepmd/npy/mixed格式,压缩度还能更高,很适合打包搬运。
deepmd -> POSCAR
接下来这部分是重头戏,讲解如何用几行代码将适用于deepmd的raw,npy格式转化为POSCAR格式。
以下代码将raw转化为POSCAR
import dpdata
Cu_dpdata = dpdata.System("Cu_bulk_deepmd", fmt="deepmd/raw")
Cu_dpdata.to(fmt="vasp/poscar", file_name="POSCAR")
或者
import dpdata
Cu_dpdata = dpdata.System("Cu_bulk_deepmd", fmt="deepmd/raw")
Cu_dpdata.to_vasp_poscar("POSCAR")
都是三行代码就能完成,完全不需要所谓的raw2pos.py。此时我们看看导出的得到的POSCAR
Cu1
1.0
2.5526554800834362e+00 -3.8564918399372628e-16 -4.9090552170547689e-16
1.2763277400417181e+00 2.2106644928618180e+00 -4.6816311855414297e-17
1.2763277400417181e+00 7.3688816428727222e-01 2.0842344717745487e+00
Cu
1
Cartesian
0.0000000000 0.0000000000 0.0000000000
这一POSCAR进行了科学计数法和Cell矩阵正则化处理,读者易证它与之前通过ASE构造的POSCAR是等价的。
从npy转到POSCAR也同理。不再赘述。
deepmd -> exyz
直接上代码。这里展示将npy格式转换为xyz格式
import dpdata
Cu_dpdata = dpdata.System("Cu_bulk_deepmd", fmt="deepmd/npy")
Cu_dpdata.to_xyz(file_name="Cu_bulk_deepmd.xyz")
得到的xyz内容:
1
Cu 0.0000000.0000000.000000
这里面是没有晶格信息的。对于有晶格信息的exyz格式,目前dpdata仅支持有Label信息(即DFT计算结果)的结构系统进行输出。
另一个输出exyz的方法见下面章节
dpdata和ase结构对象互转
我们可以将dpdata.System对象便捷地转化为ase的Atoms对象,实现结构对象的相互转化
# dpdata.System -> ase.Atoms
Cu_atoms = Cu_dpdata.to_ase_structure()[0]
# ase.Atoms -> dpdata.System
Cu_dpdata = dpdata.System().from_ase_structure(Cu_atoms)
如此即可输出结构的exyz格式
import dpdata
from ase.io import write
Cu_dpdata = dpdata.System("Cu_bulk_deepmd", fmt="deepmd/npy")
Cu_atoms = Cu_dpdata.to_ase_structure()[0]
write("Cu_bulk.exyz", Cu_atoms, format='extxyz')
此时的exyz就有晶格信息了
1
Lattice="2.5526554800834362 -3.856491839937263e-16 -4.909055217054769e-16 1.2763277400417181 2.210664492861818 -4.68163118554143e-17 1.2763277400417181 0.7368881642872722 2.0842344717745487" Properties=species:S:1:pos:R:3 pbc="T T T"
Cu 0.00000000 0.00000000 0.00000000
更快的办法:CLI
dpdata 甚至有类似于 ase 的 CLI 使用方法:
usage: dpdata [-h] [--to_file TO_FILE] [--from_format FROM_FORMAT]
[--to_format TO_FORMAT] [--no-labeled] [--multi]
[--type-map TYPE_MAP [TYPE_MAP ...]] [--version]
from_file
比如要将上面提到的deepmd/raw格式的Cu_bulk_deepmd文件夹下.raw内容转化为POSCAR,可以如此操作
dpdata --from_format deepmd/raw --no-labeled --to_file POSCAR Cu_bulk_deepmd
此操作将直接输出对应的POSCAR。和“三行代码”是一样的。
后记
dpdata里面其实有很多相关的功能,可以将结构/势能面数据进行不同格式的转化。
由于dpdata的“同化学式归为一个System或LabeledSystem,不同化学式归为一个MultiSystems”的设定,以及一系列繁多的功能,其初上手可能会有些困难,但熟练了之后会发现用起来不要太方便。而且相关开源社区活跃度高,文档完善,可以很方便地开展学习和交流。
希望大家多拥抱开源社区,少依赖祖传代码。