Nat Immunol |优质代码分享,这个热图画的太有技巧了,不仅好看,清晰,功能注释还全
- 2026-01-14 06:18:48

日常好的代码已放入免💰共享服务器中(人人皆可用):https://vip.r-py.com/
中外科学家联合绘制跨组织嗜酸性粒细胞图谱 揭示组织驻留时间驱动细胞特化新机制
近日,上海交通大学医学院、中国医学科学院系统医学研究所等中外科研团队在《自然・免疫学》(Nature Immunology)发表重磅研究成果,通过整合单细胞转录组、高维表面蛋白质组和体内命运图谱技术,成功绘制出小鼠跨组织嗜酸性粒细胞全景图谱,揭示组织驻留时间是调控嗜酸性粒细胞异质性和功能特化的核心驱动力,为免疫相关疾病研究提供了新理论框架和分子工具。

嗜酸性粒细胞作为多功能粒细胞,长期被认为与 2 型炎症、寄生虫感染相关,近年来其在代谢调控、组织修复等稳态过程中的作用逐渐被重视。然而,这类细胞在不同组织中的异质性特征及特化机制一直缺乏系统解析,且因细胞脆弱、丰度低等特性,在以往单细胞研究中常被忽略。
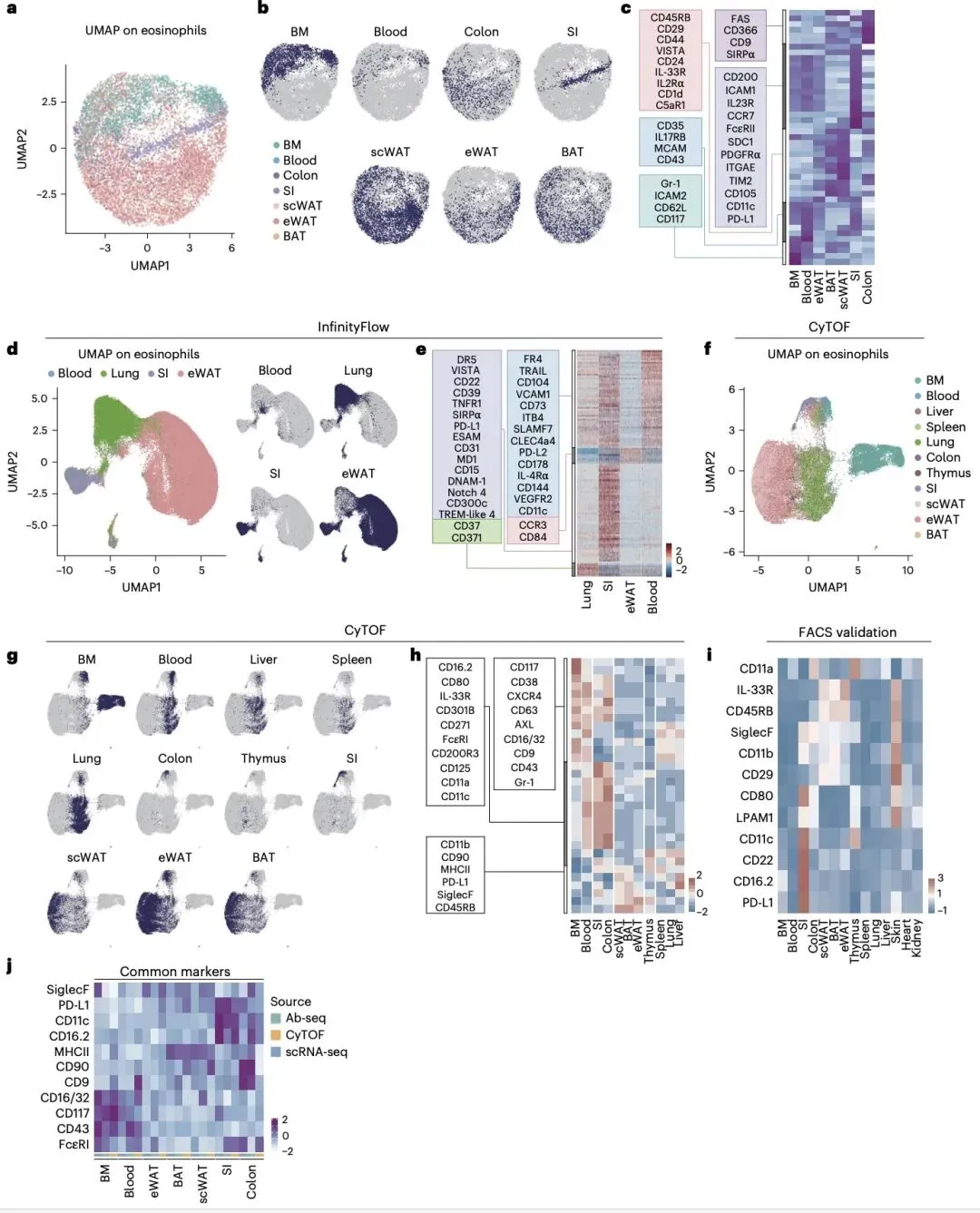
为攻克这一难题,研究团队对小鼠骨髓、血液、肺、小肠、结肠、脂肪组织等多个免疫、屏障和代谢器官进行系统采样,通过流式分选富集 CD11b+SiglecF + 嗜酸性粒细胞,结合单细胞 RNA 测序、Ab-seq、InfinityFlow 和质谱流式等多技术手段,成功鉴定出 8 种转录学不同的嗜酸性粒细胞状态,且这些状态主要按组织来源聚类。
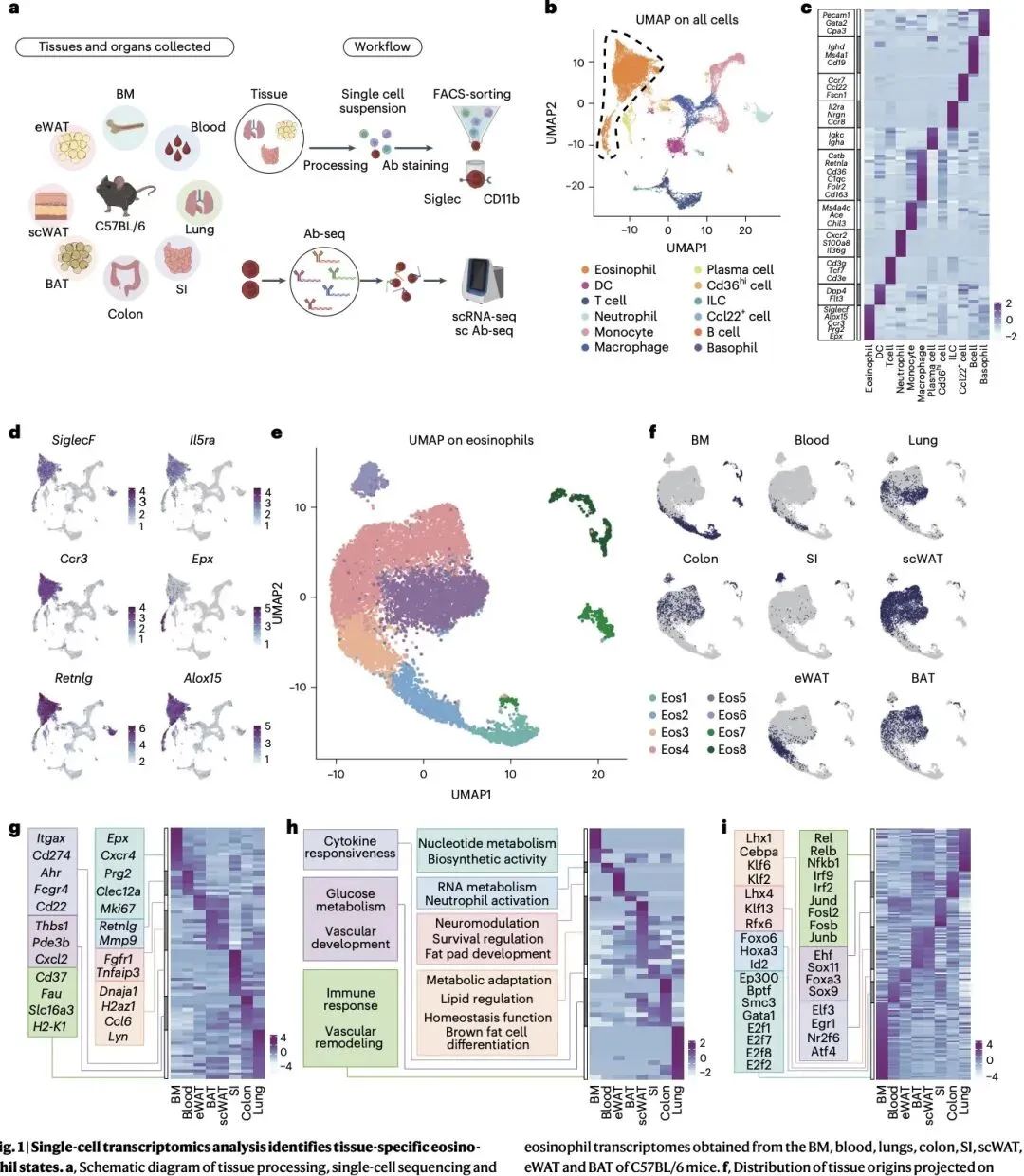
研究发现,骨髓嗜酸性粒细胞高表达增殖标志物 Mki67 和 CD371,血液中细胞则富集 CD11a 和 Mmp9,而小肠、脂肪组织等屏障和代谢组织中的嗜酸性粒细胞呈现独特的功能表型,如小肠嗜酸性粒细胞高表达 PD-L1、CD22 和 CD16.2,脂肪组织细胞则富集 IL-33R 和 CD45RB。这些组织特异性特征在转录组和蛋白质组水平高度一致,证实嗜酸性粒细胞进入组织后会发生显著的局部适应性改造。
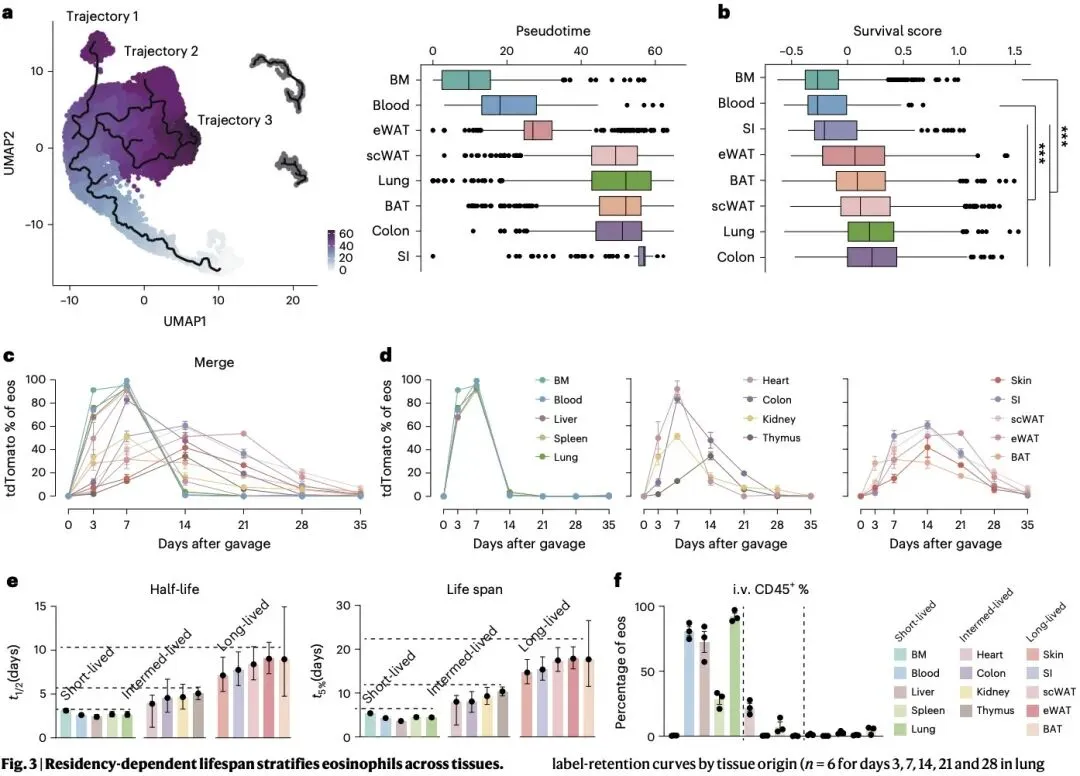
更重要的是,研究团队通过命运图谱技术首次明确了嗜酸性粒细胞的组织驻留寿命差异:肺、肝等血管丰富器官中的细胞为短寿型(<5 天),结肠细胞为中寿型(5-10 天),而小肠、脂肪组织等的细胞为长寿型(>15 天)。这种寿命差异直接关联细胞异质性程度 —— 小肠中长寿的嗜酸性粒细胞分化出 3 个转录组截然不同的亚群,分别参与代谢调控、黏膜稳态和炎症应答;而肺部短寿细胞则保持高度均一性,结肠中寿细胞异质性也相对有限。
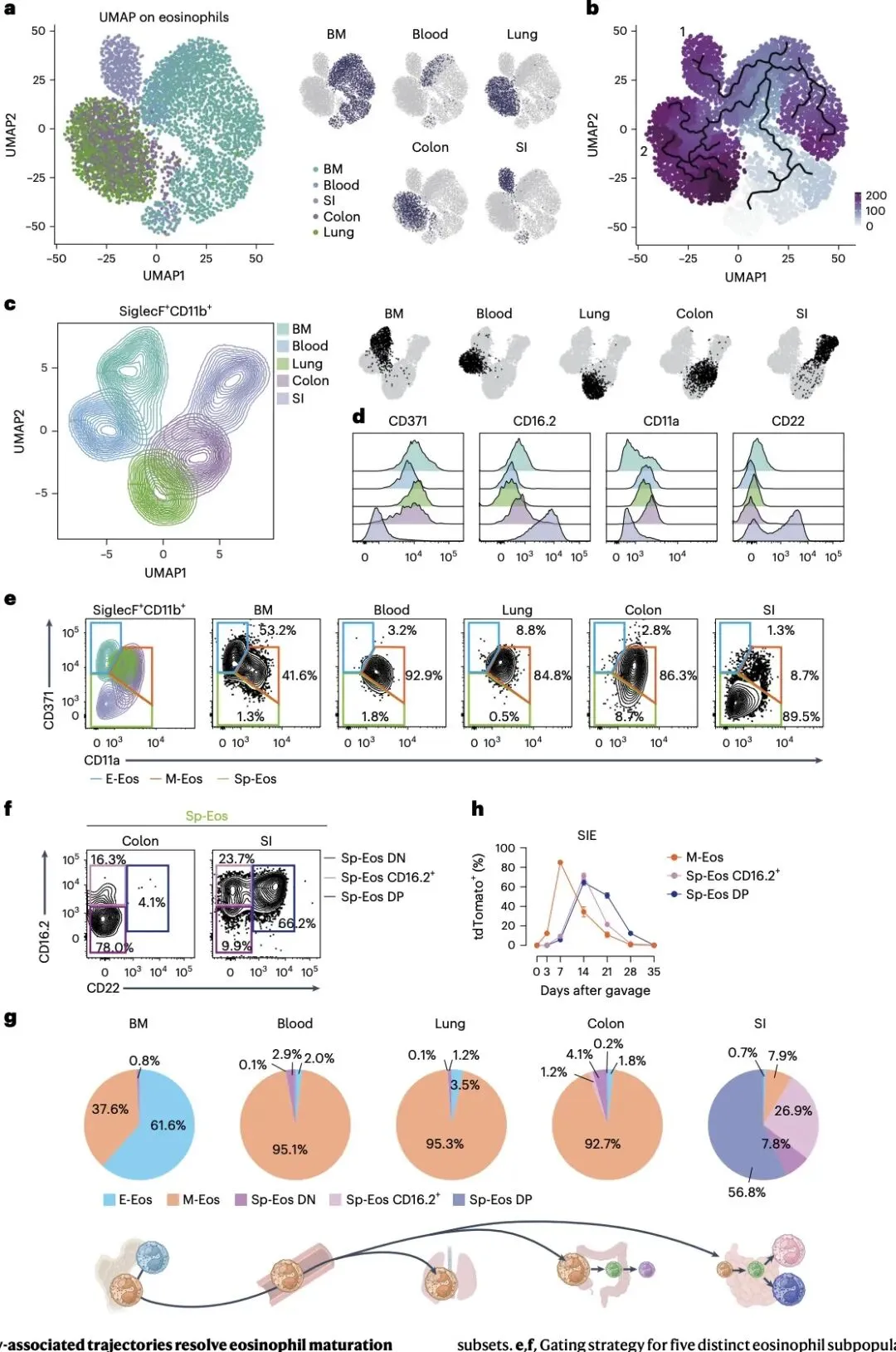
进一步的发育轨迹分析和空间定位研究显示,嗜酸性粒细胞存在从骨髓前体(E-Eos)→循环成熟型(M-Eos)→组织特化型(Sp-Eos)的连续成熟过程,CD371、CD11a、CD22 和 CD16.2 可作为区分不同成熟阶段的核心标志物。在小肠组织中,特化型嗜酸性粒细胞主要富集于绒毛区域,而成熟型细胞则局限于隐窝,这种空间分布差异进一步加剧了细胞功能分化。
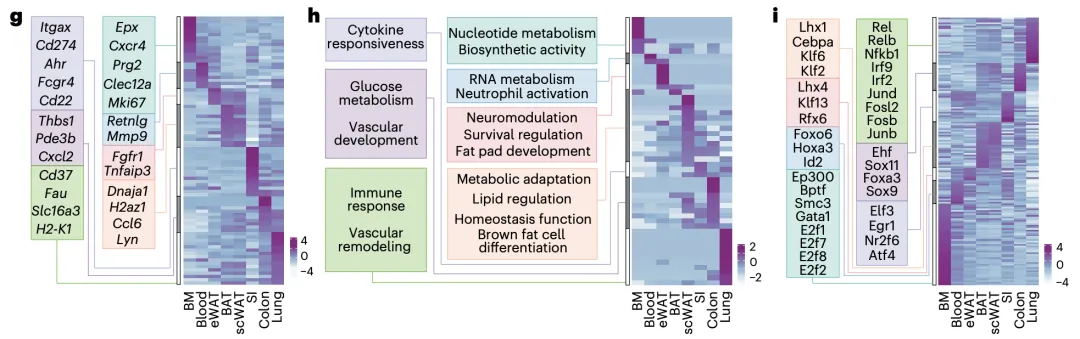
该研究还建立了 "组织驻留时间 - 细胞存活 - 功能特化" 的调控模型:组织微环境通过延长嗜酸性粒细胞驻留时间,促进其表达抗凋亡基因和功能基因,进而驱动细胞多样化。这一框架统一了此前分散的研究发现,将 CD22 + 亚群、免疫调节型亚群等多种报道的细胞类型纳入驻留时间调控体系。


library(Seurat)library(scCustomize)library(harmony)library(ggpubr)# 第一部分:基因表达矩阵构建与热图绘制(基于目标基因的组织表达水平)# 读取组织特异性新增差异基因(DEGs)数据add_gene <- read.csv("./results/tissue based add DEGs.csv")# 合并top10基因与新增差异基因,得到最终用于分析的基因集合(去重)gene_used <- union(top10, add_gene$gene)# 计算目标基因在各组织中的平均表达量# eos_0.8是单细胞RNA测序数据对象,提取RNA assay中的目标基因表达矩阵,按组织分组求均值markers_mean_exp <-aggregate(as.matrix(t(eos_0.8@assays$RNA@data[gene_used, ])), # 转置表达矩阵使基因为列、细胞为行list(Tissue = eos_0.8@meta.data[ ,"Tissue"]), # 按 Tissue 列分组mean) # 计算每组(组织)的平均表达量row.names(markers_mean_exp) <- markers_mean_exp$Tissue # 将 Tissue 列设为行名markers_mean_exp$Tissue <- c() # 删除原 Tissue 列,保留纯表达矩阵# 定义z-score标准化函数(消除基因表达量量级差异,便于跨基因比较)zscore <- function(x){return( (x - mean(x))/sd(x) ) # (原始值 - 均值) / 标准差}# 对表达矩阵按列(基因)进行z-score标准化,然后转置使基因为行、组织为列markers_plot_matrix <- apply(markers_mean_exp, 2, zscore) %>% t()# 确定每个基因表达量最高的组织(用于后续基因排序)max.avg <- apply(markers_plot_matrix, 1, which.max)# 按「基因高表达组织」分组,对基因进行排序(同组织高表达基因聚类)gene_order <- c()for(i in 1:ncol(markers_plot_matrix)){ # 遍历每个组织(列)if(sum(max.avg == i) > 1){ # 若多个基因在该组织高表达# 按该组织的表达量降序排序temp <- data.frame(gene = names(sort(markers_plot_matrix[names(max.avg)[max.avg == i],i], decreasing = T)),Tissue = colnames(markers_plot_matrix)[i], stringsAsFactors = F)}else if(sum(max.avg == i) == 1){ # 若仅1个基因在该组织高表达temp <- data.frame(gene = names(max.avg)[max.avg == i],Tissue = colnames(markers_plot_matrix)[i])}else{temp <- c() # 无基因在该组织高表达时为空}gene_order <- rbind(gene_order, temp) # 合并所有基因的排序信息}# 定义热图颜色映射(8级渐变色,基于标准化后的表达量范围)color_used <- circlize::colorRamp2(seq(min(markers_plot_matrix),max(markers_plot_matrix),length = 8),RColorBrewer::brewer.pal(9,'BuPu')[1:8])# 按排序后的基因顺序整理绘图数据plotdata <- markers_plot_matrix[gene_order$gene, ]# 绘制基因表达热图(不聚类行和列,保留手动排序结果)p <- Heatmap(plotdata, cluster_rows = FALSE, cluster_columns = FALSE,show_row_names = T, column_names_gp = gpar(fontsize = 10), # 列名(组织)字体大小row_names_gp = gpar(fontsize = 6), # 行名(基因)字体大小col = color_used, name = "Exp") # 颜色映射及图例名称# 第二部分:GO功能富集分析与通路热图绘制(对应Figure 1h)# 构建各组织差异基因列表(DEGs_BM、DEGs_Blood等为各组织预计算的差异基因数据框)csv_list <- list(BM = DEGs_BM, Blood = DEGs_Blood, eWAT = DEGs_eWAT,BAT = DEGs_BAT, scWAT = DEGs_scWAT, SI = DEGs_SI,Colon = DEGs_Colon, Lung = DEGs_Lung)# 加载功能富集分析所需包(clusterProfiler用于富集分析,org.Mm.eg.db为小鼠基因注释数据库)library(clusterProfiler)library(org.Mm.eg.db)# 准备背景基因集(所有组织差异基因的集合,转换为ENTREZID格式)all_genes <- unique(unlist(lapply(csv_list, rownames))) # 提取所有组织的差异基因符号(SYMBOL)universe_entrezid <- bitr(all_genes, fromType = "SYMBOL", # 符号转ENTREZIDtoType = c("ENTREZID"),OrgDb = org.Mm.eg.db)universe_entrezid <- unique(universe_entrezid$ENTREZID) # 去重背景基因# 筛选各组织的top200差异基因(过滤线粒体基因、核糖体基因等,调整后P值<0.05)gene_list <- list()for(tissue in names(csv_list)){ # 遍历每个组织top_gene <-csv_list[[tissue]] %>%filter(!grepl("^mt-|^Rps|^Rpl|^ENS|Rik", Symbol), # 排除线粒体、核糖体等无关基因adj.P.Val < 0.05, Grp != "others") %>% # 差异显著且非"others"分组slice_max(order_by = abs(logFC), n = 200) %>% # 按logFC绝对值取top200pull(Symbol) # 提取基因符号gene.df <- bitr(top_gene, # 符号转ENTREZIDfromType = "SYMBOL",toType = c("ENTREZID"),OrgDb = org.Mm.eg.db)gene_list[[tissue]] <- unique(gene.df$ENTREZID) # 去重后存入列表}# 批量进行GO Biological Process(BP)功能富集分析cluster_go <- compareCluster(gene_list, fun = "enrichGO",OrgDb = org.Mm.eg.db, ont = "BP", # 小鼠数据库,BP本体universe = universe_entrezid, # 背景基因集pvalueCutoff = 0.05, # P值阈值qvalueCutoff = 0.05, # 校正后P值阈值readable = TRUE, pool = TRUE) # 输出基因符号,合并富集结果# 提取富集分析结果并转换为数据框BP_result<-as.data.frame(cluster_go@compareClusterResult)# (可选)从文件读取预计算的富集结果(避免重复分析)BP_result<-read.csv("./results/BP_compareCluster_results.csv",row.names = 1)# 读取手动筛选的关键通路列表(根据研究重点选择的BP通路)pathway<-read.csv("./results/chosen BP pathway new.csv")pathway_used <- pathway %>%pull(Description) %>%unique() # 提取通路描述并去重# 整理通路富集热图数据(按组织×通路整理校正后P值的-log10转换值)heatmap_df <- BP_result %>%filter(Description %in% pathway_used) %>% # 仅保留筛选后的通路dplyr::select(Cluster, Description, p.adjust) %>% # 选择组织、通路、校正后P值mutate(logp = -log10(p.adjust)) %>% # 转换为-log10(p.adjust)(值越大富集越显著)dplyr::select(-p.adjust) %>% # 删除原P值列pivot_wider(names_from = Cluster, values_from = logp, values_fill = 0) %>% # 宽格式转换tibble::column_to_rownames(var = "Description") # 通路设为行名# 定义组织顺序(按研究设计的逻辑顺序排列)Tissue_order <- c("BM","Blood","eWAT","BAT","scWAT","SI","Colon","Lung")heatmap_df <- heatmap_df[ ,Tissue_order] # 按顺序调整列# 对通路富集矩阵按行(通路)进行z-score标准化markers_plot_matrix <- apply(heatmap_df, 1, zscore) %>% t()# 确定每个通路富集最显著的组织(用于通路排序)max.avg <- apply(markers_plot_matrix, 1, which.max)# 按「通路富集最显著组织」分组,对通路进行排序(同组织富集通路聚类)pathway_order <- c()for(i in 1:ncol(markers_plot_matrix)){ # 遍历每个组织(列)if(sum(max.avg == i) > 1){ # 若多个通路在该组织富集显著# 按该组织的富集分数降序排序temp <- data.frame(pathway = names(sort(markers_plot_matrix[names(max.avg)[max.avg == i],i], decreasing = T)),tissue = colnames(markers_plot_matrix)[i], stringsAsFactors = F)}else if(sum(max.avg == i) == 1){ # 若仅1个通路在该组织富集显著temp <- data.frame(pathway = names(max.avg)[max.avg == i],tissue = colnames(markers_plot_matrix)[i])}else{temp <- c() # 无通路在该组织富集显著时为空}pathway_order <- rbind(pathway_order, temp) # 合并所有通路的排序信息}pathway_order <- pathway_order[nrow(pathway_order):1,] # 反转顺序(可选,调整绘图布局)pathways_labeled <- pathway_used # 通路标签# 定义通路热图颜色映射(同基因表达热图)color_used <- circlize::colorRamp2(seq(min(markers_plot_matrix),max(markers_plot_matrix),length = 8),RColorBrewer::brewer.pal(9,'BuPu')[1:8])# 按排序后的通路顺序整理绘图数据plotdata_BP<-as.data.frame(markers_plot_matrix[pathway_order$pathway,])# 绘制通路富集热图(不聚类行和列,保留手动排序结果)p <- Heatmap(as.matrix(plotdata_BP),cluster_rows = FALSE,cluster_columns = FALSE,show_row_names = T,column_names_gp = gpar(fontsize = 10), # 组织列名字体大小row_names_gp = gpar(fontsize = 9), # 通路行名字体大小row_names_max_width = max_text_width(rownames(plotdata_BP)), # 适配长通路名称col = color_used,show_heatmap_legend = FALSE) # 隐藏图例(可根据需要调整为TRUE)
(我们自己开发的服务器,很多意想不到的功能
免💰服务器地址:https://vip.r-py.com/)
生信钱同学团队提供生信便捷式“云服务器”、“数据分析”、“生信学习班”以及“生信交流群”等内容,有需要者可以加我:sx_qtx01
相关推文内容:
今天开课了,没服务器直接送——多组学与机器学习联合分析(机器学习分析代谢组、蛋白组、宏基因组、网络药理学、转录组课)
学生信当然要与时俱进了,零基础也能学的AI生信课(AI助力生信入门班即将开始(AI课)
超多生信内容学习,感兴趣可以了解下(单细胞课程)
没有服务器,单细胞数据搞不定?看看我们做出来的结果包含啥?我们目前做好了这些pipeline,可以帮你做(数据分析) 公共共享服务器已开启,无门槛尽管用,不限制资源(免💰服务器) 今晚 7 点开始,12节线上课,不收费,把开源的QuPath病理和荧光图像分析的技巧学明白 手把手复现Nature的生信教学即将开始,感兴趣的可以了解下 当我同学用这个套路发了10几篇SCI,并拿了国奖,我惊呆了——文献计量学+AI大语言模型学习(文献计量培训) Gemini详细安装教程,自动分析数据,不是器械式写代码,而是帮你做好,就是这么智能(AI自动写代码,自动分析数据)