利用python进行多序列比对可视化
- 2026-01-14 10:56:06
利用python进行多序列比对可视化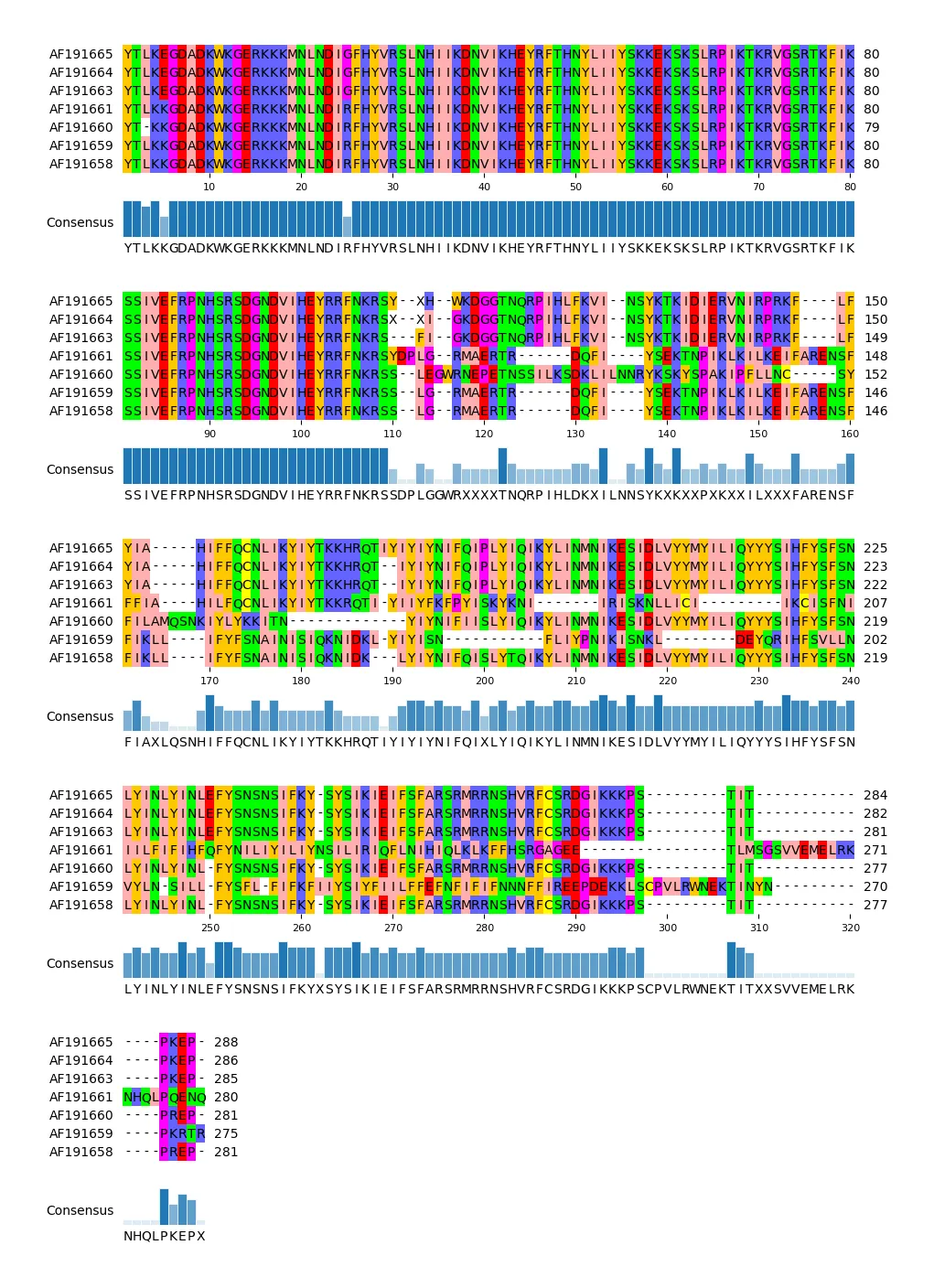

利用python进行多序列比对可视化
我们在找到相似序列时候,会碰到一个问题,就是如何对序列进行可视化,这里笔者推荐使用python模块pymsaviz 进行多序列比对可视化。

进行多序列比对
多序列比对有多种方式可以进行,这里使用mafft进行,示例数据为rpl16.pro.fa。
在conda中安装mafft后,进行下列操作,生成msa文件。
mafft --maxiterate 1000 --thread 10 rpl16.fa > rpl16.msa.fa
msa为多序列比对的结果,其本质依然是fasta文件, 只不过,多了一些内容。

上图为msa的结果,可以发现两个序列最后对齐,少的地方用—填补。
利用pymsaviz进行序列比对可视化
安装
conda install -c conda-forge -c bioconda pymsaviz
实际操作
from pymsaviz import MsaViz
mv = MsaViz(r"E:\markdown\多序列比对可视化\rpl16.pro.msa.fa", wrap_length=80, show_count=True,show_consensus=True)
mv.plotfig()
其中wrap_length为每一行所展示的氨基酸的个数
show_count,展示每行氨基酸数目
show_consensus,展示每一列所含有的最多的氨基酸,即底下的柱状图。
结果如下:

进阶操作
from pymsaviz import MsaViz
mv = MsaViz(r"E:\markdown\多序列比对可视化\rpl16.pro.msa.fa", wrap_length=80, show_count=True,show_consensus=True)
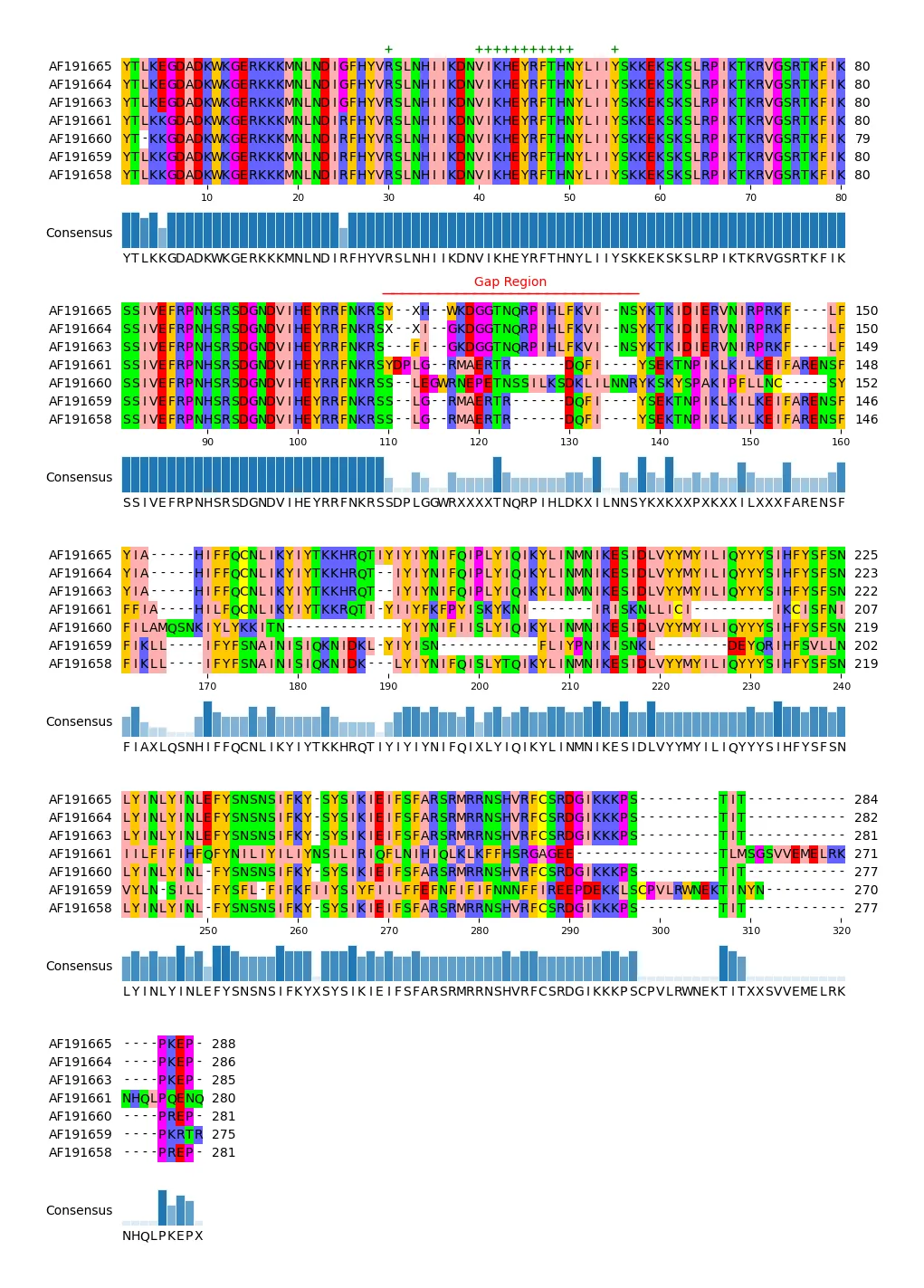
##在30 40-50 以及55 处打上标记
mv.add_markers([30, (40, 50), 55], color="green", marker="+")
##在110到113加上注释
mv.add_text_annotation((110, 113), "Gap Region", text_color="red", range_color="red")
mv.plotfig()

总结
pymsaviz作为一个简单的多序列比对小工具,是十分简单以及高效的,但是要注意,pymsaviz对于核苷酸并不是十分友好,建议大家只使用氨基酸序列。
请联系我们
笔者也创建一个数据分析交流群,可以扫描二维码添加笔者联系方式,拉你入群,笔者作为群主会不定时发放福利,另外群内含有众多大佬,不定时分享就业以及科研经验解决你的生信分析难题。
推荐阅读
本文来自网友投稿或网络内容,如有侵犯您的权益请联系我们删除,联系邮箱:wyl860211@qq.com 。
