使用自己Windows系统电脑简易搭建Linux生信分析平台
- 2026-02-02 05:38:58
使用自己Windows系统电脑简易搭建Linux生信分析平台鉴于如今越来越多的分析算法或工具仅面向 Linux 用户开放,而且租赁服务器的费用高昂,还存在数据泄露的风险,因此在自己设备上搭建 一个Linux分析平台显得尤为重要。在这里,针对QTLMR用户需求,特在QTLMR包使用帮助文档——一网打尽Post GWAS分析(←点击前往)中更新了简易安装视频教程,完成电脑子系统Ubuntu的安装,耗时约10余分钟。 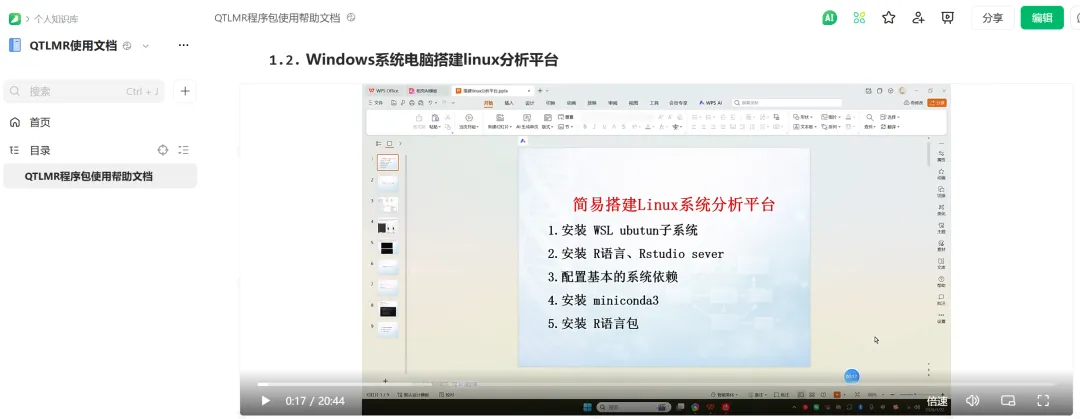
安装完成后,就可以使用自己的电脑登录Linux端口的Rstudio server,运行R语言代码了。这样就可以在自己的设备上进行MiXeR、csMR、finemapping、CELLECT等等系列分析啦...当然,基础好点的同学,也可以用来做单细胞等其它生信分析~ 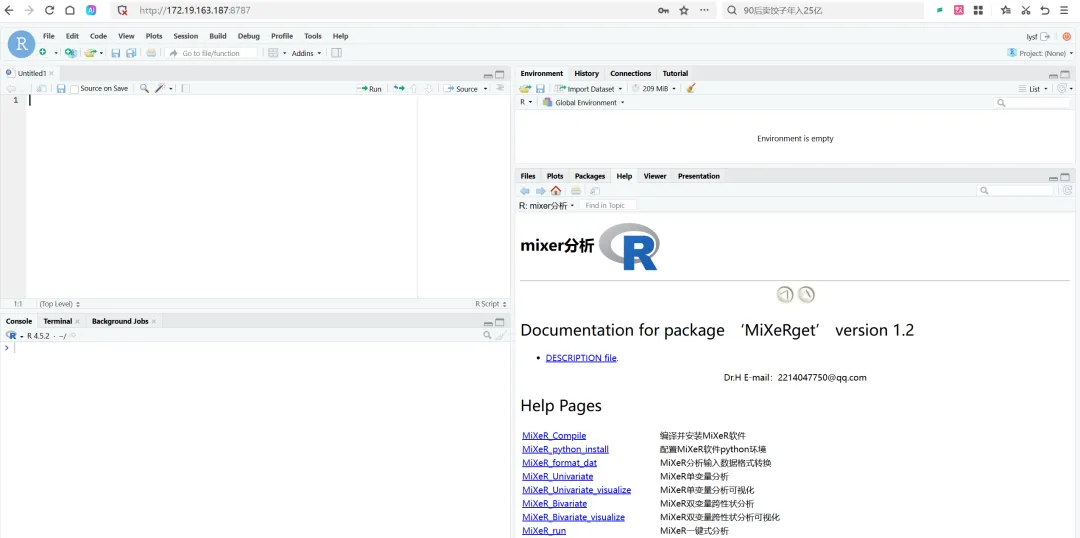
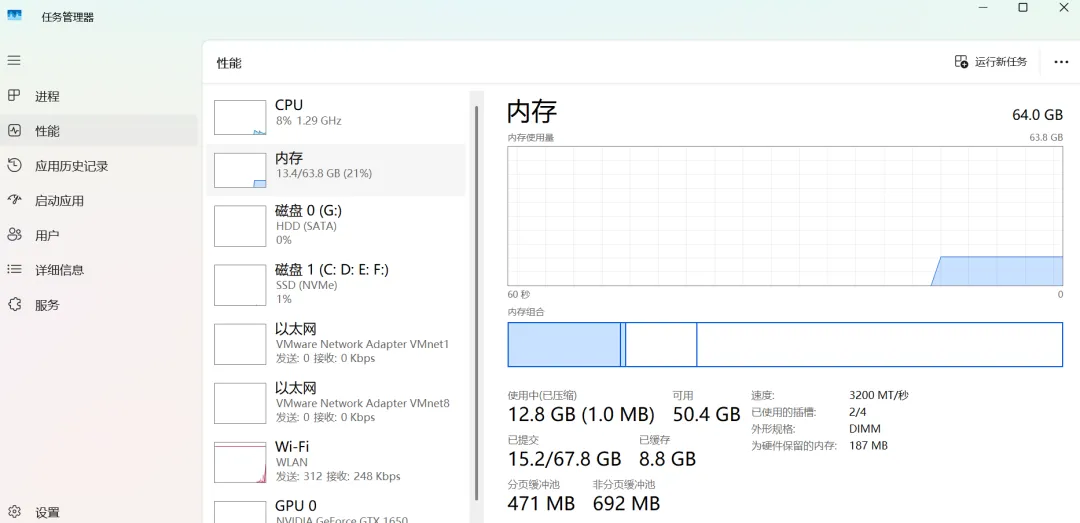
分析的速率,取决于设备配置的硬件性能,与计算资源的分配。多大的能力办多大的事,分析的时候可以看看任务管理器资源的调动情况。如果内存、CPU等资源不够时,可能需要大家自行去购买硬件,自行扩展。后续有时间再教大家如何部署juterpy notebook,用于python分析...大家自行尝试一下吧~ 
最后,建议基础较差的小伙伴稍微去学习一下Linux基础,凡事问AI不是解决问题的根本办法,也会严重拖累工作效率...在B站上有很多免费公开的基础课程,花几天时间就能学习完成,有一定基础后,工作起来就更加顺心。毕竟AI不是万能的~ 如果您对Post GWAS方法感兴趣,可以使用QTLMR包进行分析,QTLMR是一个功能强大的R语言包,整合了MR、gsMap、coloc、hyprcoloc、SuSiE、Finemaping、PWcoco、SMR、MTAG、Hess、METAL、LDSC、HDL、Popcorn、LOGODetect、SUPERGNOVA、GNOVA、CPASSOC、PLACO、MiXeR、cond/conjFDR、FM_summary、 GCTA_fastBAT、MAGMA、MR-JTI、SMulTiXcan、S-PrediXcan、PoPs、FLAMES、FOCUS、FUSION、MTWAS及UTMOST分析等多种算法及多种可视化绘图功能,详细可见QTLMR R语言程序包 (←点击前往)及QTLMR包使用帮助文档——一网打尽Post GWAS分析(←点击前往)。 最后,大家可以关注下微信公众号 更多内容将不断更新...




本文来自网友投稿或网络内容,如有侵犯您的权益请联系我们删除,联系邮箱:wyl860211@qq.com 。
