【生存分析机器学习】Python04-随机生存森林模型RSF及可视化
- 2026-01-31 01:16:38


概念:基于机器学习的生存分析方法,使用多个生存树集成。
原理:通过Bootstrap抽样建树,节点分裂使用log-rank统计量最大化生存差异。累积风险函数使用Nelson-Aalen估计器。
思想:无需假设风险比例,处理复杂交互和非线性关系。
应用:高维数据生存预测,如医学研究中的预后模型。
可视化:变量重要性图(VIMP);最小深度图;部分依赖图。
公共卫生意义:在大数据公共卫生中识别复杂风险模式,用于个性化预防。

-数据预处理:
-模型构建:
-训练:
-评估:
-可视化:
-保存结果:
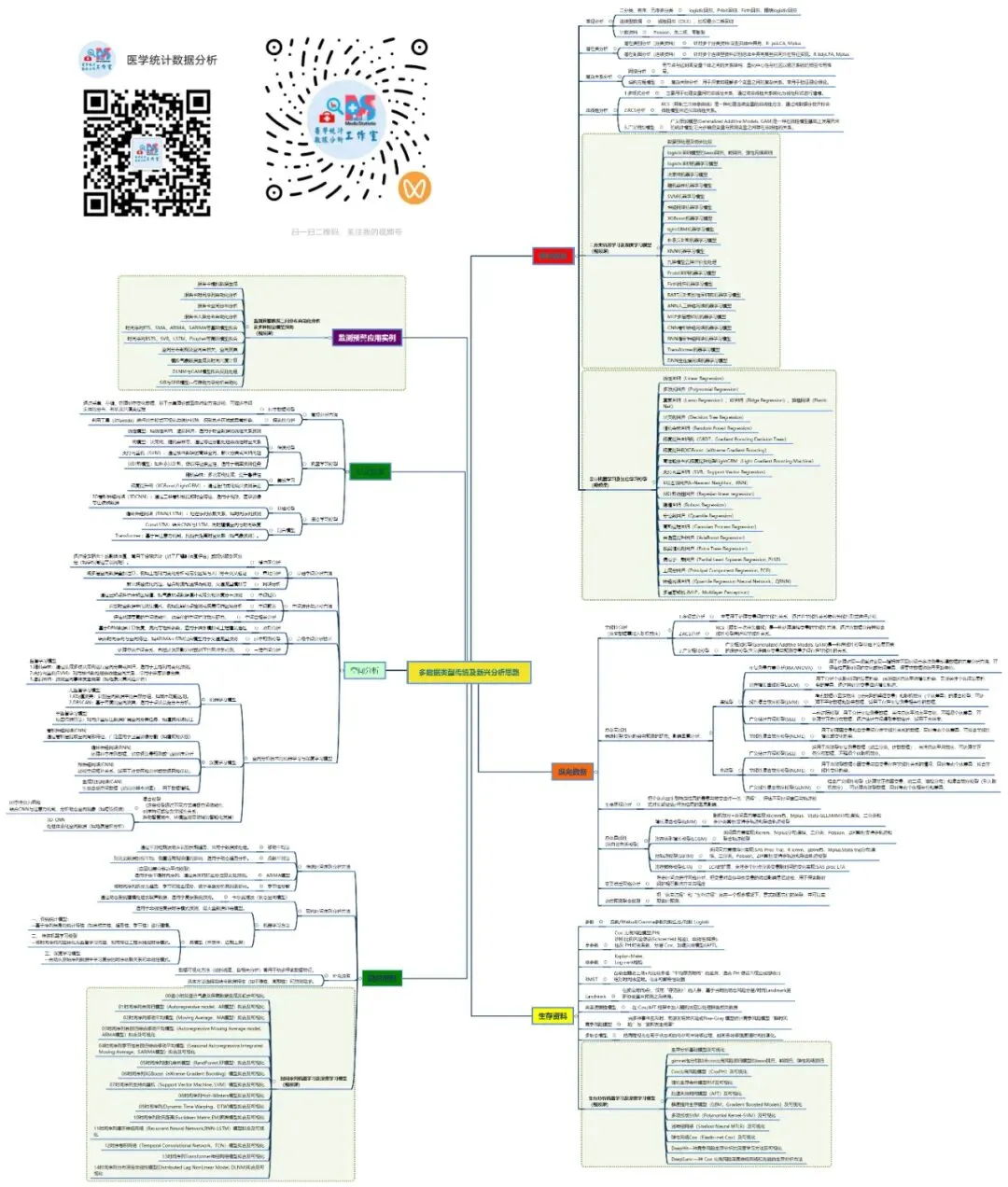
生存分析机器学习模型主要分为传统统计扩展模型、树基集成模型和深度学习模型三大类,它们通过不同机制处理删失数据并预测事件发生时间。评价方法则聚焦于区分能力、校准效果和临床实用性,核心指标包括一致性指数(C-index)、Brier分数和时间依赖ROC曲线等。
# pip install pandas numpy matplotlib seaborn scikit-learn scikit-survival lifelinesimport osimport pandas as pdimport numpy as npimport matplotlib.pyplot as pltimport seaborn as snsfrom sklearn.model_selection import train_test_splitfrom sklearn.preprocessing import StandardScaler, LabelEncoderfrom sklearn.metrics import roc_curve, aucfrom sklearn.calibration import calibration_curvefrom sklearn.inspection import permutation_importancefrom sksurv.ensemble import RandomSurvivalForestfrom sksurv.util import Survfrom sksurv.metrics import concordance_index_censoredfrom sksurv.metrics import cumulative_dynamic_aucfrom sksurv.metrics import brier_scoreimport scipy.stats as statsimport warningswarnings.filterwarnings('ignore')# 设置中文字体和输出目录plt.rcParams['font.sans-serif'] = ['SimHei', 'Microsoft YaHei']plt.rcParams['axes.unicode_minus'] = False# 创建结果文件夹desktop_path = os.path.join(os.path.expanduser("~"), "Desktop")results_dir = os.path.join(desktop_path, "Results模型-rsf-comprehensive")os.makedirs(results_dir, exist_ok=True)# 1. 数据准备和预处理def load_and_preprocess_data():""" 加载和预处理数据 """ try:# 尝试读取实际数据 data = pd.read_excel(os.path.join(desktop_path, "示例数据.xlsx"), sheet_name="示例数据")print("成功读取示例数据") except:# 创建模拟数据print("未找到示例数据,创建模拟数据...") np.random.seed(2025) n_samples = 500 data = pd.DataFrame({'时间': np.random.exponential(100, n_samples),'结局': np.random.choice([0, 1], n_samples, p=[0.7, 0.3]),'指标1': np.random.normal(50, 10, n_samples),'指标2': np.random.normal(25, 5, n_samples),'指标3': np.random.normal(100, 20, n_samples),'指标4': np.random.normal(10, 2, n_samples),'指标5': np.random.normal(75, 15, n_samples),'指标6': np.random.normal(30, 6, n_samples),'指标7': np.random.normal(60, 12, n_samples),'指标8': np.random.choice(['A', 'B', 'C'], n_samples),'肥胖程度': np.random.choice(['正常', '超重', '肥胖'], n_samples),'教育水平': np.random.choice(['小学', '中学', '大学'], n_samples),'血型': np.random.choice(['A', 'B', 'O', 'AB'], n_samples) })# 确保时间变量为正数 data['时间'] = np.abs(data['时间'])# 数据预处理 data['时间'] = pd.to_numeric(data['时间'], errors='coerce') data['结局'] = pd.to_numeric(data['结局'], errors='coerce') data['结局'] = data['结局'].fillna(0).astype(int)# 处理分类变量 - 使用独热编码 categorical_cols = ['指标8', '肥胖程度', '教育水平', '血型']for col in categorical_cols:if col in data.columns:# 使用独热编码 dummies = pd.get_dummies(data[col], prefix=col) data = pd.concat([data, dummies], axis=1) data = data.drop(columns=[col])# 删除缺失值 data = data.dropna()print(f"数据维度: {data.shape}")print(f"结局分布:\n{data['结局'].value_counts()}")return data# 2. 描述性统计分析def descriptive_analysis(data, results_dir):"""生成描述性统计分析"""print("\n=== 描述性统计分析 ===")# 数值变量描述 numeric_cols = data.select_dtypes(include=[np.number]).columns numeric_cols = [col for col in numeric_cols if col not in ['时间', '结局']] desc_stats = data[numeric_cols].describe().T desc_stats['missing'] = data[numeric_cols].isnull().sum()# 按结局分组的描述if len(data['结局'].unique()) > 1: grouped_stats = data.groupby('结局')[numeric_cols].mean()# 保存结果 desc_stats.to_csv(os.path.join(results_dir, "数值变量描述性统计.csv"))return desc_stats# 3. 划分训练集和测试集def split_data(data, test_size=0.3, random_state=2025):"""划分训练集和测试集""" train_data, test_data = train_test_split( data, test_size=test_size, random_state=random_state, stratify=data['结局'] )print(f"训练集样本数: {len(train_data)}")print(f"测试集样本数: {len(test_data)}")return train_data, test_data# 4. 构建随机生存森林模型def build_rsf_model(train_data, duration_col='时间', event_col='结局'):"""构建随机生存森林模型"""print("\n=== 构建随机生存森林模型 ===")# 选择数值型预测变量 numeric_cols = train_data.select_dtypes(include=[np.number]).columns predictor_cols = [col for col in numeric_cols if col not in [duration_col, event_col]]
# 5. 模型性能评估def evaluate_model(rsf, train_data, test_data, predictor_cols, X_train, y_train, results_dir):"""评估模型性能"""print("\n=== 模型性能评估 ===")# 准备测试数据 X_test = test_data[predictor_cols].values y_test = np.array([(bool(row['结局']), row['时间'])for _, row in test_data.iterrows()], dtype=[('event', '?'), ('time', '<f8')])# 训练集C-index train_pred = rsf.predict(X_train) train_cindex = concordance_index_censored(y_train['event'], y_train['time'], train_pred)[0]# 测试集C-index test_pred = rsf.predict(X_test) test_cindex = concordance_index_censored(y_test['event'], y_test['time'], test_pred)[0]print(f"训练集C-index: {train_cindex:.3f}")print(f"测试集C-index: {test_cindex:.3f}")# 时间依赖性ROC曲线 time_points = [12, 24, 36] # 1年、2年、3年 auc_values = calculate_time_dependent_auc(rsf, X_test, y_test, time_points, results_dir)# Brier分数 brier_scores = calculate_brier_score(rsf, test_data, X_test, time_points, results_dir)return {'train_cindex': train_cindex,'test_cindex': test_cindex,'auc_values': auc_values,'brier_scores': brier_scores,'test_pred': test_pred,'X_test': X_test,'y_test': y_test }def calculate_time_dependent_auc(rsf, X_test, y_test, time_points, results_dir):"""计算时间依赖性AUC"""print("\n=== 时间依赖性AUC计算 ===")# 使用scikit-survival的cumulative_dynamic_auc函数 try:# 获取风险分数 risk_scores = rsf.predict(X_test)# 计算时间依赖性AUC auc_values = {}for time_point in time_points:# 使用cumulative_dynamic_auc计算特定时间点的AUC# 注意:这里我们使用一个时间范围,然后取最接近我们目标时间点的值times = np.percentile(y_test['time'], np.linspace(5, 95, 20)) iauc, mean_auc = cumulative_dynamic_auc(y_test, y_test, risk_scores, times)# 找到最接近目标时间点的索引 time_idx = np.argmin(np.abs(times - time_point)) auc_value = iauc[time_idx] auc_values[time_point] = auc_valueprint(f"{time_point}个月AUC: {auc_value:.3f}") except Exception as e:print(f"使用cumulative_dynamic_auc计算AUC时出错: {e}")print("使用替代方法计算AUC...")# 替代方法:使用生存函数计算 auc_values = calculate_auc_alternative(rsf, X_test, y_test, time_points)return auc_valuesdef calculate_auc_alternative(rsf, X_test, y_test, time_points):"""替代方法计算AUC""" auc_values = {}# 获取生存函数 surv_funcs = rsf.predict_survival_function(X_test)for time_point in time_points:# 计算在特定时间点的风险分数(1-生存概率) risk_scores = []for sf in surv_funcs:# 找到最接近的时间点times = sf.x time_idx = np.argmin(np.abs(times - time_point)) risk_scores.append(1 - sf.y[time_idx]) risk_scores = np.array(risk_scores)# 创建二元标签 y_true_binary = (y_test['time'] <= time_point) & y_test['event']if y_true_binary.sum() > 0: # 确保有阳性样本 fpr, tpr, _ = roc_curve(y_true_binary, risk_scores) roc_auc = auc(fpr, tpr) auc_values[time_point] = roc_aucprint(f"{time_point}个月AUC: {roc_auc:.3f}")else:print(f"{time_point}个月: 无事件发生,无法计算AUC") auc_values[time_point] = np.nanreturn auc_valuesdef calculate_brier_score(rsf, test_data, X_test, time_points, results_dir):"""计算Brier分数"""print("\n=== Brier分数计算 ===") brier_scores = {}# 获取生存函数 surv_funcs = rsf.predict_survival_function(X_test)for time_point in time_points:# 找到最接近的时间点times = surv_funcs[0].x time_idx = np.argmin(np.abs(times - time_point))# 计算在该时间点的生存概率 surv_probs = []for sf in surv_funcs: surv_probs.append(sf.y[time_idx]) surv_probs = np.array(surv_probs)# 计算观察到的结果 observed = ((test_data['时间'] <= time_point) & (test_data['结局'] == 1)).astype(int)# 计算Brier分数 brier_score = np.mean((observed - (1 - surv_probs)) ** 2) brier_scores[time_point] = brier_scoreprint(f"{time_point}个月Brier分数: {brier_score:.3f}")# 保存Brier分数 brier_df = pd.DataFrame(list(brier_scores.items()), columns=['Time', 'Brier_Score']) brier_df.to_csv(os.path.join(results_dir, "Brier分数.csv"), index=False)return brier_scores# 6. 可视化分析def create_visualizations(rsf, train_data, test_data, predictor_cols, performance_metrics, results_dir):"""创建所有可视化图表"""print("\n=== 创建可视化图表 ===")# 准备测试数据 X_test = performance_metrics['X_test'] y_test = performance_metrics['y_test']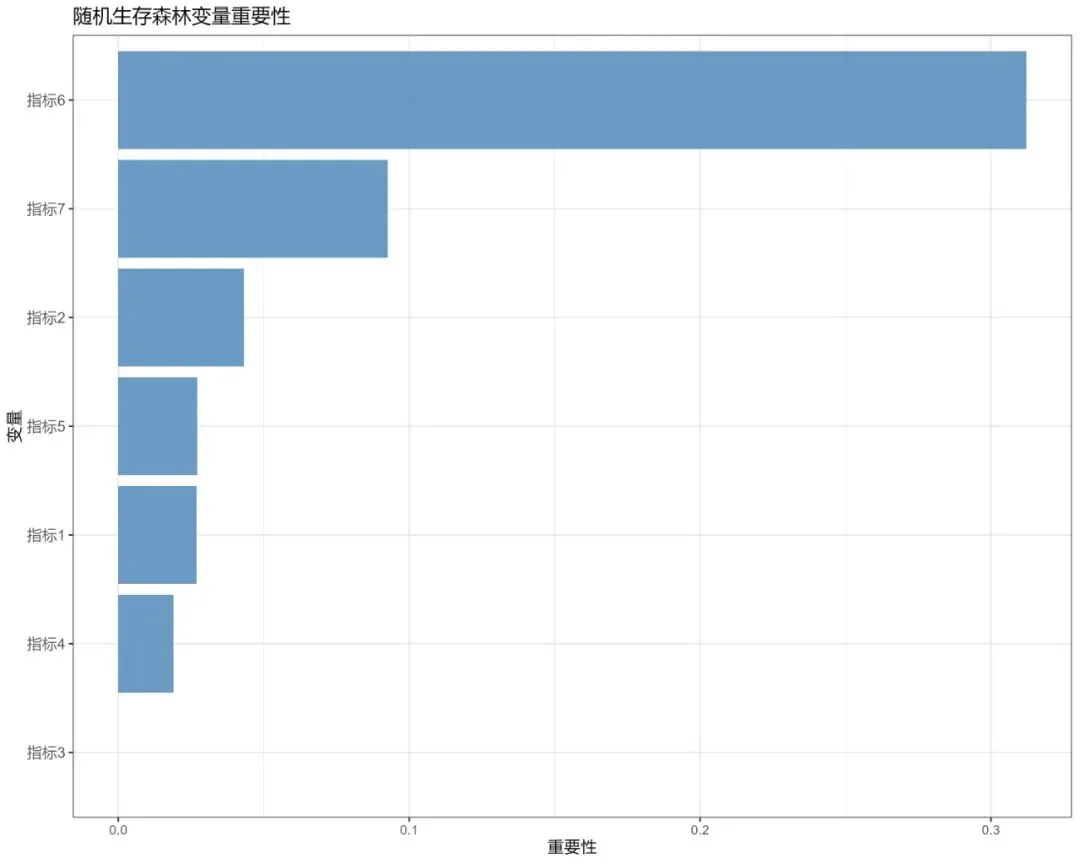
# 6.1 变量重要性图 plot_variable_importance(rsf, X_test, y_test, predictor_cols, results_dir)# 6.2 生存曲线 plot_survival_curves(rsf, X_test, results_dir)# 6.3 校准曲线 plot_calibration_curve(rsf, test_data, X_test, results_dir)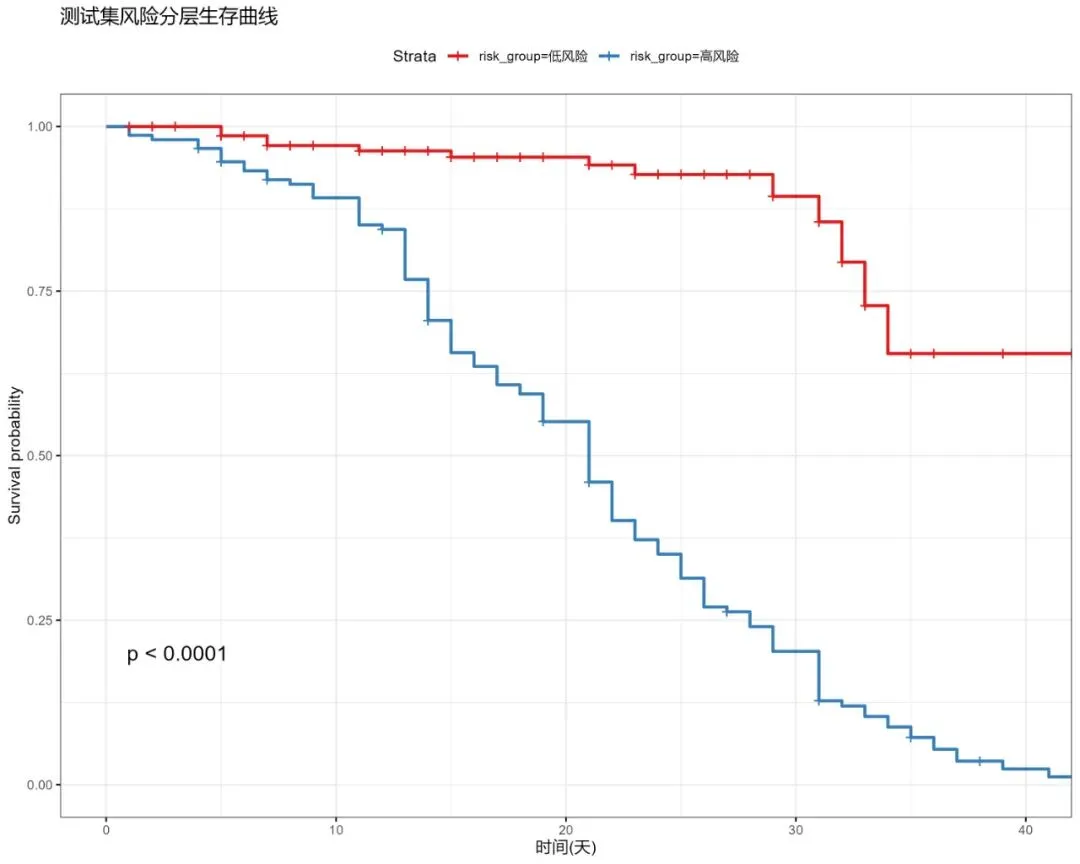
# 6.4 风险分层可视化 plot_risk_stratification(test_data, performance_metrics['test_pred'], results_dir)# 6.5 临床决策曲线分析 plot_decision_curve_analysis(test_data, performance_metrics['test_pred'], results_dir)# 6.6 时间依赖性ROC曲线 plot_time_dependent_roc(rsf, X_test, y_test, results_dir)def plot_variable_importance(rsf, X_test, y_test, predictor_cols, results_dir):"""绘制变量重要性图 - 使用排列重要性"""print("计算变量重要性...") try:# 使用排列重要性 result = permutation_importance( rsf, X_test, y_test, n_repeats=10, random_state=2025, n_jobs=-1 ) feature_importance = result.importances_mean# 创建重要性数据框 importance_df = pd.DataFrame({'Variable': predictor_cols,'Importance': feature_importance }).sort_values('Importance', ascending=True)# 绘制水平条形图 plt.figure(figsize=(12, 8)) plt.barh(importance_df['Variable'], importance_df['Importance'], color='steelblue', alpha=0.8) plt.xlabel('排列重要性') plt.title('随机生存森林变量重要性') plt.grid(True, alpha=0.3)# 添加数值标签for i, v in enumerate(importance_df['Importance']): plt.text(v + 0.001, i, f'{v:.3f}', va='center') plt.tight_layout() plt.savefig(os.path.join(results_dir, "变量重要性图.jpg"), dpi=300, bbox_inches='tight') plt.close()# 保存变量重要性数据 importance_df.to_csv(os.path.join(results_dir, "变量重要性.csv"), index=False)print("变量重要性图创建成功") except Exception as e:print(f"计算变量重要性时出错: {e}")# 创建简单的变量重要性图(基于特征名称) plt.figure(figsize=(12, 8)) plt.barh(range(len(predictor_cols)), [1] * len(predictor_cols), color='steelblue', alpha=0.8) plt.yticks(range(len(predictor_cols)), predictor_cols) plt.xlabel('重要性 (使用排列重要性计算失败)') plt.title('随机生存森林变量重要性') plt.grid(True, alpha=0.3) plt.tight_layout() plt.savefig(os.path.join(results_dir, "变量重要性图.jpg"), dpi=300, bbox_inches='tight') plt.close()def plot_survival_curves(rsf, X_test, results_dir):"""绘制生存曲线""" plt.figure(figsize=(12, 8))# 获取生存函数 surv_funcs = rsf.predict_survival_function(X_test)# 绘制前50个样本的生存曲线(避免过于拥挤) n_samples_to_plot = min(50, len(surv_funcs))for i in range(n_samples_to_plot): plt.plot(surv_funcs[i].x, surv_funcs[i].y, alpha=0.3, color='blue')# 绘制平均生存曲线 all_times = [] all_surv_probs = []for sf in surv_funcs: all_times.extend(sf.x) all_surv_probs.extend(sf.y)# 计算分位数曲线 unique_times = np.unique(all_times) surv_probs_at_times = []for time_point in unique_times: probs_at_time = []for sf in surv_funcs: idx = np.argmin(np.abs(sf.x - time_point)) probs_at_time.append(sf.y[idx]) surv_probs_at_times.append(np.median(probs_at_time)) plt.plot(unique_times, surv_probs_at_times, 'r-', linewidth=3, label='中位生存曲线') plt.xlabel('时间') plt.ylabel('生存概率') plt.title('随机生存森林生存曲线') plt.legend() plt.grid(True, alpha=0.3) plt.tight_layout() plt.savefig(os.path.join(results_dir, "生存曲线.jpg"), dpi=300, bbox_inches='tight') plt.close()def plot_calibration_curve(rsf, test_data, X_test, results_dir):"""绘制校准曲线""" plt.figure(figsize=(10, 8)) time_points = [12, 24, 36] colors = ['#4DAF4A', '#984EA3', '#FF7F00']# 获取生存函数 surv_funcs = rsf.predict_survival_function(X_test)for i, time_point in enumerate(time_points):# 找到最接近的时间点times = surv_funcs[0].x time_idx = np.argmin(np.abs(times - time_point))# 计算在该时间点的生存概率 pred_probs = []for sf in surv_funcs: pred_probs.append(1 - sf.y[time_idx]) # 转换为风险概率 pred_probs = np.array(pred_probs)# 计算观察到的风险 observed_risk = ((test_data['时间'] <= time_point) & (test_data['结局'] == 1)).astype(int)# 创建校准数据 prob_true, prob_pred = calibration_curve( observed_risk, pred_probs, n_bins=10, strategy='quantile' ) plt.plot(prob_pred, prob_true, 'o-', color=colors[i], label=f'{time_point}个月', linewidth=2, markersize=6) plt.plot([0, 1], [0, 1], '--', color='gray', label='理想校准') plt.xlabel('预测风险概率') plt.ylabel('观察风险概率') plt.title('随机生存森林 - 校准曲线') plt.legend() plt.grid(True, alpha=0.3) plt.tight_layout() plt.savefig(os.path.join(results_dir, "校准曲线.jpg"), dpi=300, bbox_inches='tight') plt.close()def plot_risk_stratification(test_data, test_pred, results_dir):"""风险分层可视化""" plt.figure(figsize=(12, 8))# 根据预测风险中位数分层 risk_median = np.median(test_pred) test_data = test_data.copy() test_data['risk_group'] = np.where(test_pred > risk_median, '高风险', '低风险')# 使用lifelines绘制分层生存曲线 from lifelines import KaplanMeierFitter kmf = KaplanMeierFitter()for group in ['低风险', '高风险']: group_data = test_data[test_data['risk_group'] == group] kmf.fit(group_data['时间'], group_data['结局'], label=group) kmf.plot_survival_function() plt.xlabel('时间') plt.ylabel('生存概率') plt.title('测试集风险分层生存曲线') plt.grid(True, alpha=0.3) plt.tight_layout() plt.savefig(os.path.join(results_dir, "风险分层生存曲线.jpg"), dpi=300, bbox_inches='tight') plt.close()def plot_decision_curve_analysis(test_data, test_pred, results_dir):"""临床决策曲线分析""" plt.figure(figsize=(10, 8)) thresholds = np.linspace(0.01, 0.99, 50) net_benefits = []for threshold in thresholds:# 根据阈值决定治疗 risk_threshold = np.quantile(test_pred, threshold) treat_by_model = test_pred > risk_threshold true_positives = np.sum(treat_by_model & (test_data['结局'] == 1)) false_positives = np.sum(treat_by_model & (test_data['结局'] == 0)) n = len(test_data) net_benefit = (true_positives / n) - (false_positives / n) * (threshold / (1 - threshold)) net_benefits.append(net_benefit) plt.plot(thresholds, net_benefits, 'b-', linewidth=2, label='模型') plt.axhline(y=0, color='gray', linestyle='--', label='不治疗')# 所有治疗策略 event_rate = test_data['结局'].mean() treat_all_benefit = [event_rate - (1 - event_rate) * (t / (1 - t)) for t in thresholds] plt.plot(thresholds, treat_all_benefit, 'r--', linewidth=2, label='全部治疗') plt.xlabel('决策阈值') plt.ylabel('标准化净获益') plt.title('随机生存森林 - 临床决策曲线分析 (DCA)') plt.legend() plt.grid(True, alpha=0.3) plt.tight_layout() plt.savefig(os.path.join(results_dir, "临床决策曲线.jpg"), dpi=300, bbox_inches='tight') plt.close()def plot_time_dependent_roc(rsf, X_test, y_test, results_dir):"""绘制时间依赖性ROC曲线""" plt.figure(figsize=(10, 8)) time_points = [12, 24, 36] colors = ['#4DAF4A', '#984EA3', '#FF7F00']# 获取生存函数 surv_funcs = rsf.predict_survival_function(X_test)for i, time_point in enumerate(time_points):# 计算在特定时间点的风险分数(1-生存概率) risk_scores = []for sf in surv_funcs:# 找到最接近的时间点times = sf.x time_idx = np.argmin(np.abs(times - time_point)) risk_scores.append(1 - sf.y[time_idx]) risk_scores = np.array(risk_scores)# 创建二元标签 y_true_binary = (y_test['time'] <= time_point) & y_test['event']if y_true_binary.sum() > 0: # 确保有阳性样本 fpr, tpr, _ = roc_curve(y_true_binary, risk_scores) roc_auc = auc(fpr, tpr) plt.plot(fpr, tpr, color=colors[i], linewidth=2, label=f'{time_point}个月 (AUC = {roc_auc:.2f})') plt.plot([0, 1], [0, 1], 'k--', alpha=0.5, label='随机分类') plt.xlabel('1 - 特异度') plt.ylabel('敏感度') plt.title('随机生存森林 - 时间依赖性ROC曲线') plt.legend() plt.grid(True, alpha=0.3) plt.tight_layout() plt.savefig(os.path.join(results_dir, "时间ROC曲线.jpg"), dpi=300, bbox_inches='tight') plt.close()# 7. 保存结果和生成报告def save_results_and_report(rsf, performance_metrics, train_data, test_data, predictor_cols, results_dir):"""保存所有结果并生成报告"""print("\n=== 保存结果和生成报告 ===")# 7.1 保存模型性能指标 performance_df = pd.DataFrame([ ['训练集C-index', performance_metrics['train_cindex']], ['测试集C-index', performance_metrics['test_cindex']], ] + [[f'{time_point}个月AUC', auc_value]for time_point, auc_value in performance_metrics['auc_values'].items()], columns=['Metric', 'Value'] ) performance_df.to_csv(os.path.join(results_dir, "模型性能指标.csv"), index=False)# 7.2 生成文本报告 generate_text_report(performance_metrics, len(train_data), len(test_data), results_dir)# 7.3 保存工作空间 import joblib joblib.dump({'model': rsf,'performance_metrics': performance_metrics,'train_data': train_data,'test_data': test_data,'predictor_cols': predictor_cols }, os.path.join(results_dir, "分析环境.pkl"))def generate_text_report(performance_metrics, n_train, n_test, results_dir):"""生成文本报告""" report_content = f"""随机生存森林模型综合分析报告================================数据概览--------训练集样本数: {n_train}测试集样本数: {n_test}模型性能指标-----------训练集C-index: {performance_metrics['train_cindex']:.3f}测试集C-index: {performance_metrics['test_cindex']:.3f}时间依赖性AUC值:"""for time_point, auc_value in performance_metrics['auc_values'].items(): report_content += f"{time_point}个月AUC: {auc_value:.3f}\n" report_content += f"""Brier分数:"""for time_point, brier_score in performance_metrics['brier_scores'].items(): report_content += f"{time_point}个月Brier分数: {brier_score:.3f}\n" report_content += f"""关键结论--------模型在测试集的区分能力(C-index): {performance_metrics['test_cindex']:.3f}平均Brier分数: {np.mean(list(performance_metrics['brier_scores'].values())):.3f}"""# 性能问题提示if performance_metrics['test_cindex'] < 0.5: report_content += """!!! 警告: 测试集C-index低于0.5,模型在测试集上表现不佳 !!!可能原因:- 训练集和测试集分布差异大- 模型参数需要调整- 数据预处理问题建议检查数据划分的随机性和数据质量。""" with open(os.path.join(results_dir, "综合分析报告.txt"), "w", encoding='utf-8') as f: f.write(report_content)# 主函数def main():print("开始随机生存森林模型综合分析...")# 1. 数据准备和预处理 data = load_and_preprocess_data()# 2. 描述性统计分析 desc_stats = descriptive_analysis(data, results_dir)# 3. 划分训练集和测试集 train_data, test_data = split_data(data)# 4. 构建随机生存森林模型 rsf, predictor_cols, X_train, y_train = build_rsf_model(train_data)# 5. 模型性能评估 performance_metrics = evaluate_model(rsf, train_data, test_data, predictor_cols, X_train, y_train, results_dir)# 6. 可视化分析 create_visualizations(rsf, train_data, test_data, predictor_cols, performance_metrics, results_dir)# 7. 保存结果和生成报告 save_results_and_report(rsf, performance_metrics, train_data, test_data, predictor_cols, results_dir)print(f"\n=== 随机生存森林模型综合分析已完成 ===")print(f"所有结果已保存到 {results_dir} 文件夹中。")print("包含的性能评价指标:")print("- 综合区分能力 (C-index)")print("- 时间依赖性ROC曲线 (AUC)")print("- Brier分数 (校准度)")print("- 临床决策曲线 (DCA)")print("- 风险分层分析")print("- 变量重要性分析")print(f"\n关键指标:")print(f"训练集C-index: {performance_metrics['train_cindex']:.3f}")print(f"测试集C-index: {performance_metrics['test_cindex']:.3f}")print(f"时间点AUC值: {', '.join([f'{auc_value:.3f}' for auc_value in performance_metrics['auc_values'].values()])}")print(f"平均Brier分数: {np.mean(list(performance_metrics['brier_scores'].values())):.3f}")if __name__ == "__main__": main()🔍随机生存森林模型及可视化
概念:随机生存森林是一种基于集成学习(Bagging)的机器学习方法,通过构建多棵生存树并综合其结果进行预测。
原理:使用Bootstrap重抽样生成多个训练集。为每棵树随机选择特征进行节点分裂,分裂准则通常是最大化子节点间的生存差异(如Log-rank统计量)。综合所有树的结果,例如通过Nelson-Aalen估计器计算累积风险函数,最终聚合得到整体预测。
思想:通过模型平均提高预测精度,并能有效捕捉变量间的复杂交互作用和非线性效应,且不依赖于比例风险等假设。
应用:适用于高维临床数据的生存预测和变量重要性排序,例如利用患者多维度指标预测其长期生存情况。
可视化:变量重要性图:展示每个变量对模型预测准确性的贡献程度(基于置换重要性或VIMP)。最小深度图:反映变量在树中首次被用作分裂点的平均深度,有助于识别关键变量。
公共卫生意义:能够从大规模健康数据(如电子健康记录)中挖掘影响社区人群健康的复杂因素,助力慢性病风险预测和分层管理。

医学统计数据分析分享交流SPSS、R语言、Python、ArcGis、Geoda、GraphPad、数据分析图表制作等心得。承接数据分析,论文返修,医学统计,机器学习,生存分析,空间分析,问卷分析业务。若有投稿和数据分析代做需求,可以直接联系我,谢谢!
“医学统计数据分析”公众号右下角;
找到“联系作者”,
可加我微信,邀请入粉丝群!

有临床流行病学数据分析
如(t检验、方差分析、χ2检验、logistic回归)、
(重复测量方差分析与配对T检验、ROC曲线)、
(非参数检验、生存分析、样本含量估计)、
(筛检试验:灵敏度、特异度、约登指数等计算)、
(绘制柱状图、散点图、小提琴图、列线图等)、
机器学习、深度学习、生存分析
等需求的同仁们,加入【临床】粉丝群。
疾控,公卫岗位的同仁,可以加一下【公卫】粉丝群,分享生态学研究、空间分析、时间序列、监测数据分析、时空面板技巧等工作科研自动化内容。
有实验室数据分析需求的同仁们,可以加入【生信】粉丝群,交流NCBI(基因序列)、UniProt(蛋白质)、KEGG(通路)、GEO(公共数据集)等公共数据库、基因组学转录组学蛋白组学代谢组学表型组学等数据分析和可视化内容。
或者可扫码直接加微信进群!!!



精品视频课程-“医学统计数据分析”视频号付费合集

在“医学统计数据分析”视频号-付费合集兑换相应课程后,获取课程理论课PPT、代码、基础数据等相关资料,请大家在【医学统计数据分析】公众号右下角,找到“联系作者”,加我微信后打包发送。感谢您的支持!!
