Gromacs模拟后分析代码实操
- 2026-02-03 17:11:17
Gromacs模拟后分析代码实操
①导出去除水的轨迹以及蛋白文件 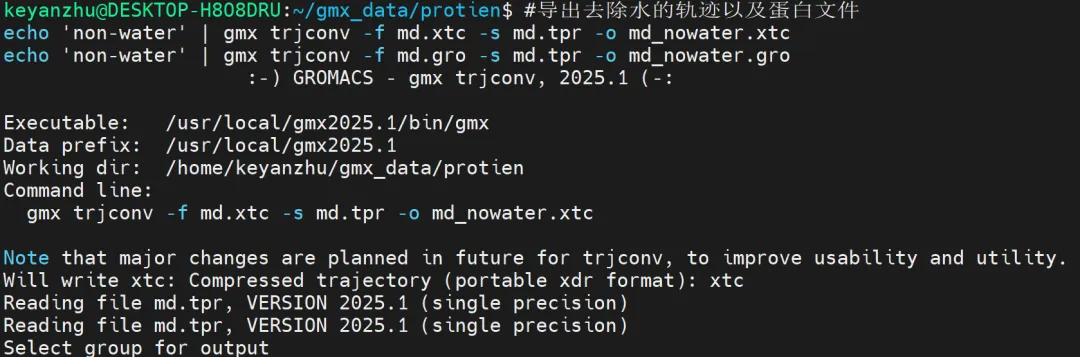
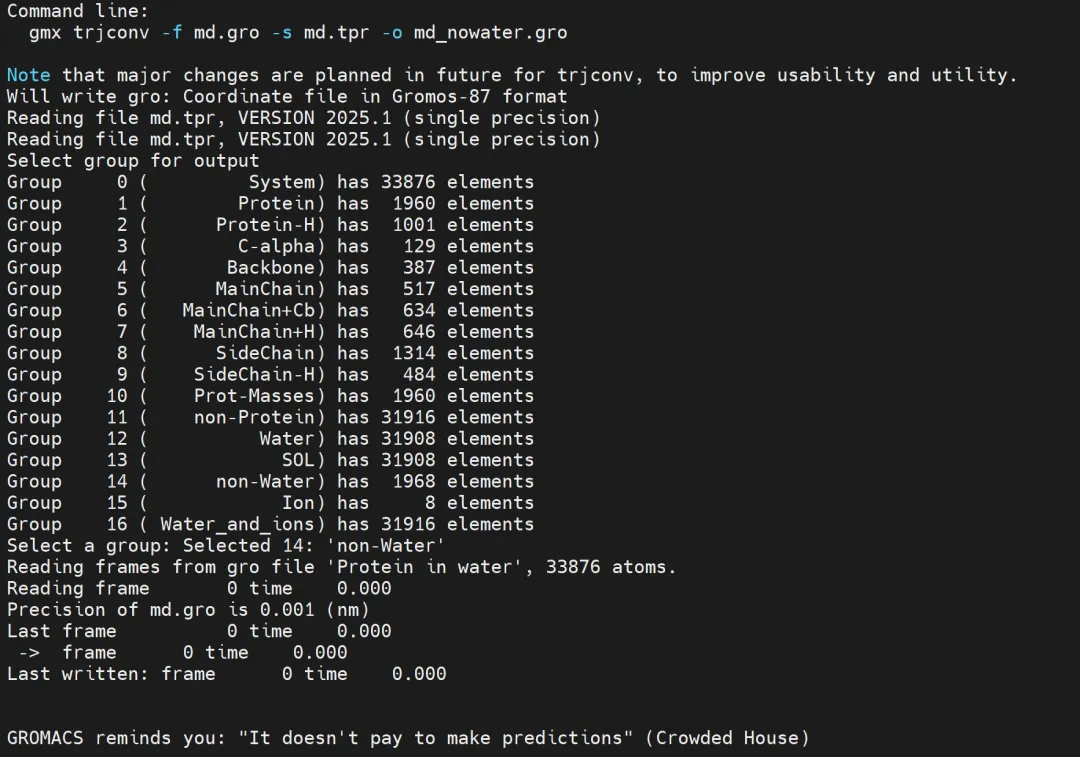
产生去掉水的轨迹以及结构文件 
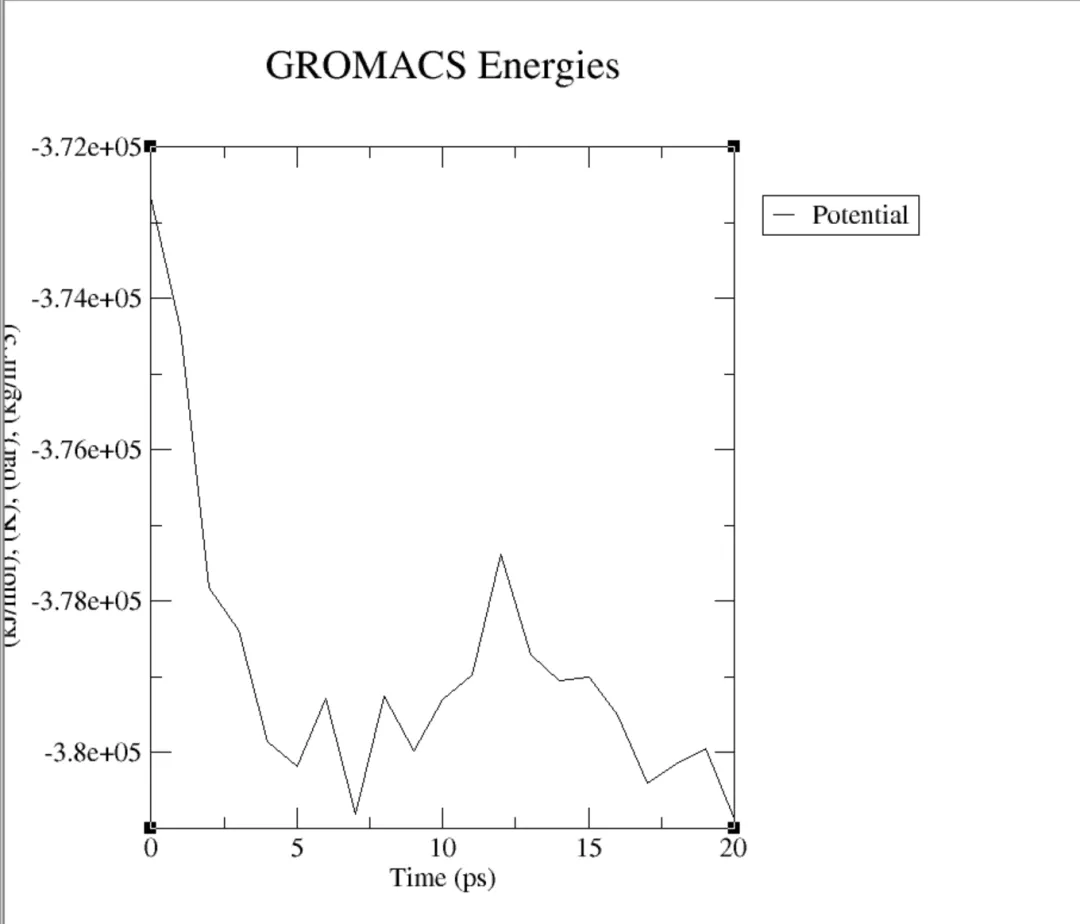
②查看能量温度压力密度变化等 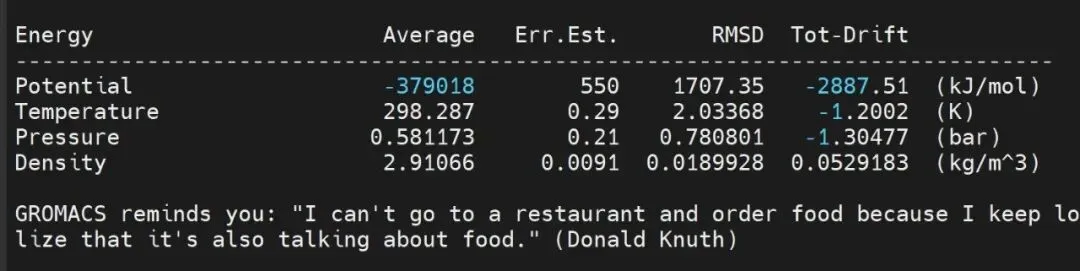
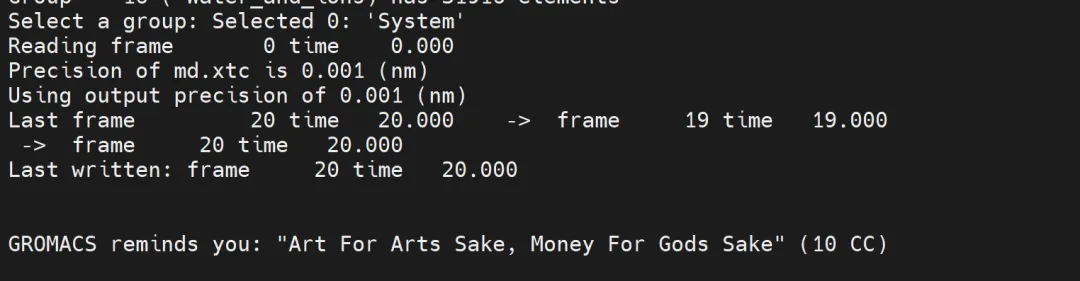
能量信息相关的xvg文件以及 noPBC轨迹文件,同时利用xmgrace可以简单查看曲线,如果没有安装可以通过sudo apt install grace安装 
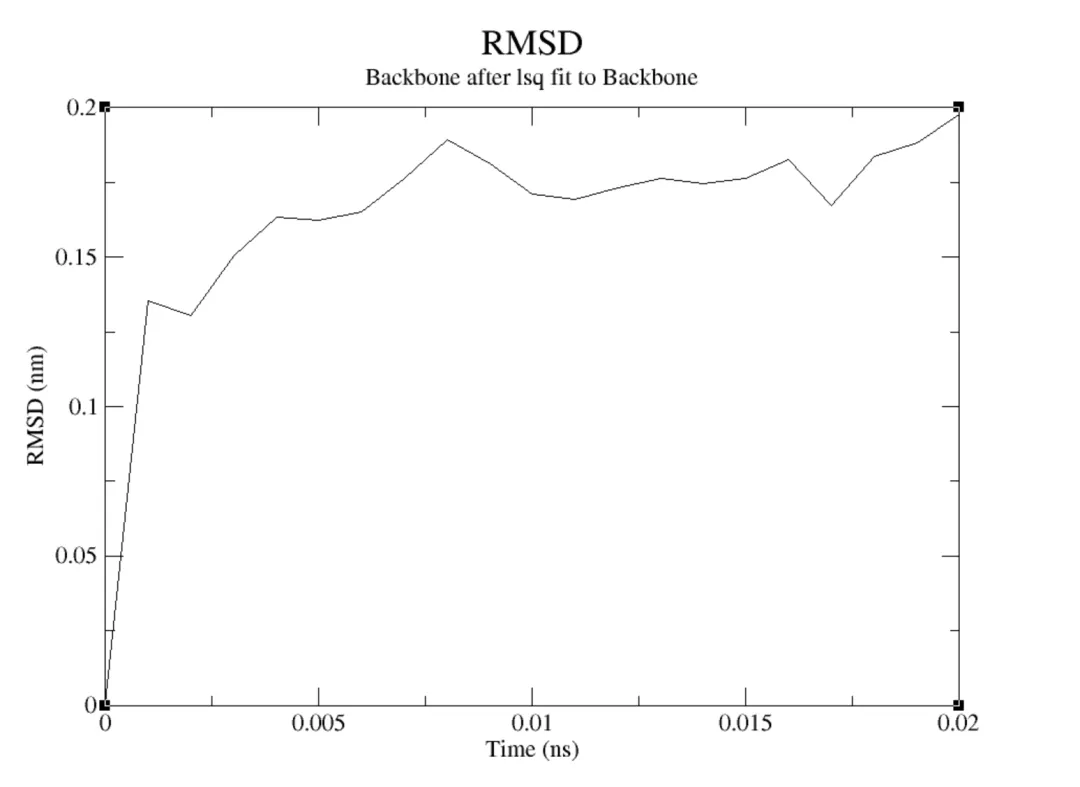
③RMSD 
rmsd.xvg的文件 
④RMSF 
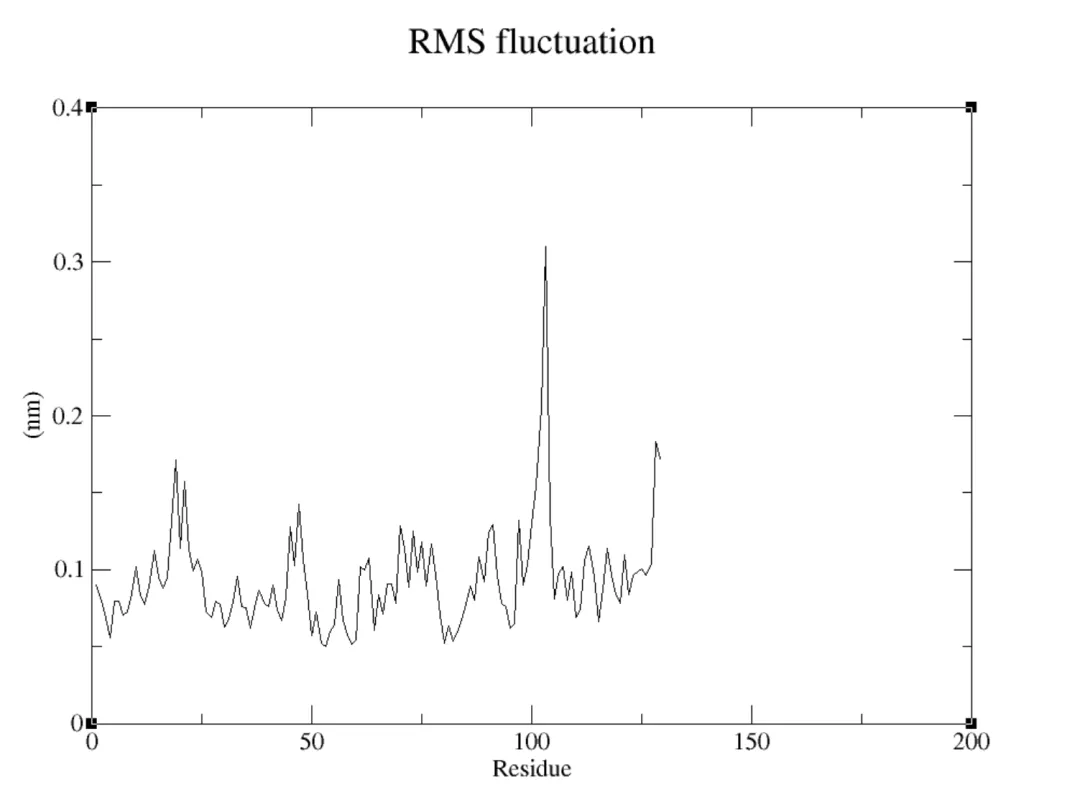
应该有 
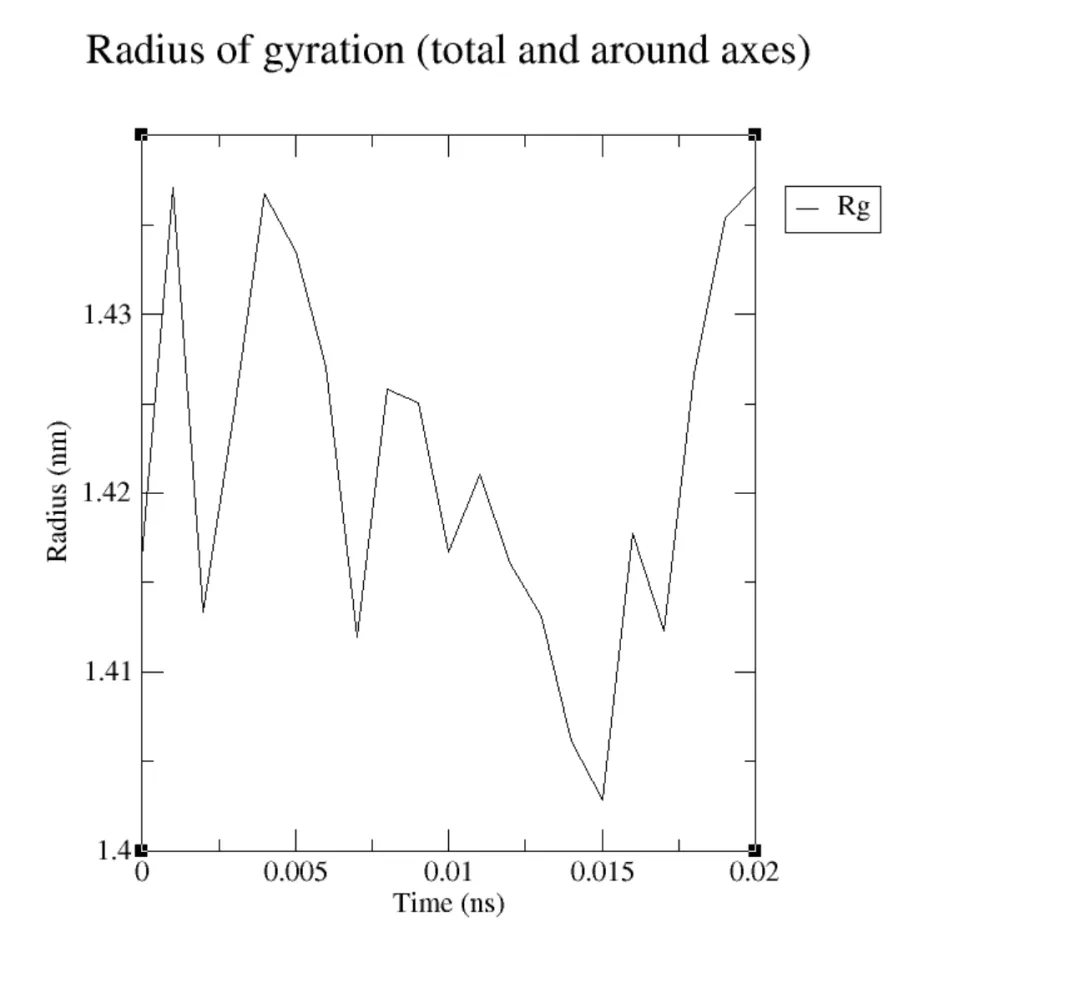
⑤RG 
⑥二级结构 
Xmgrace查看注意加上参数-nxy 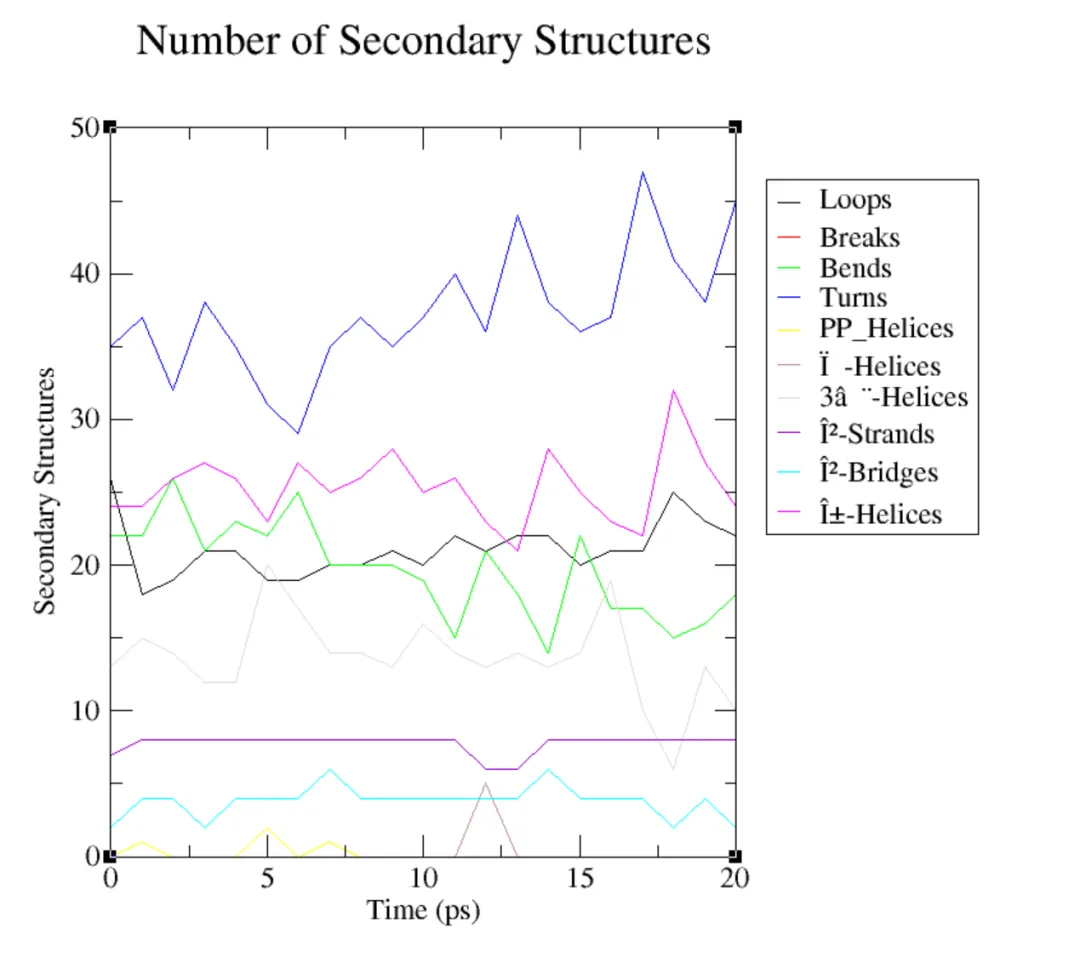
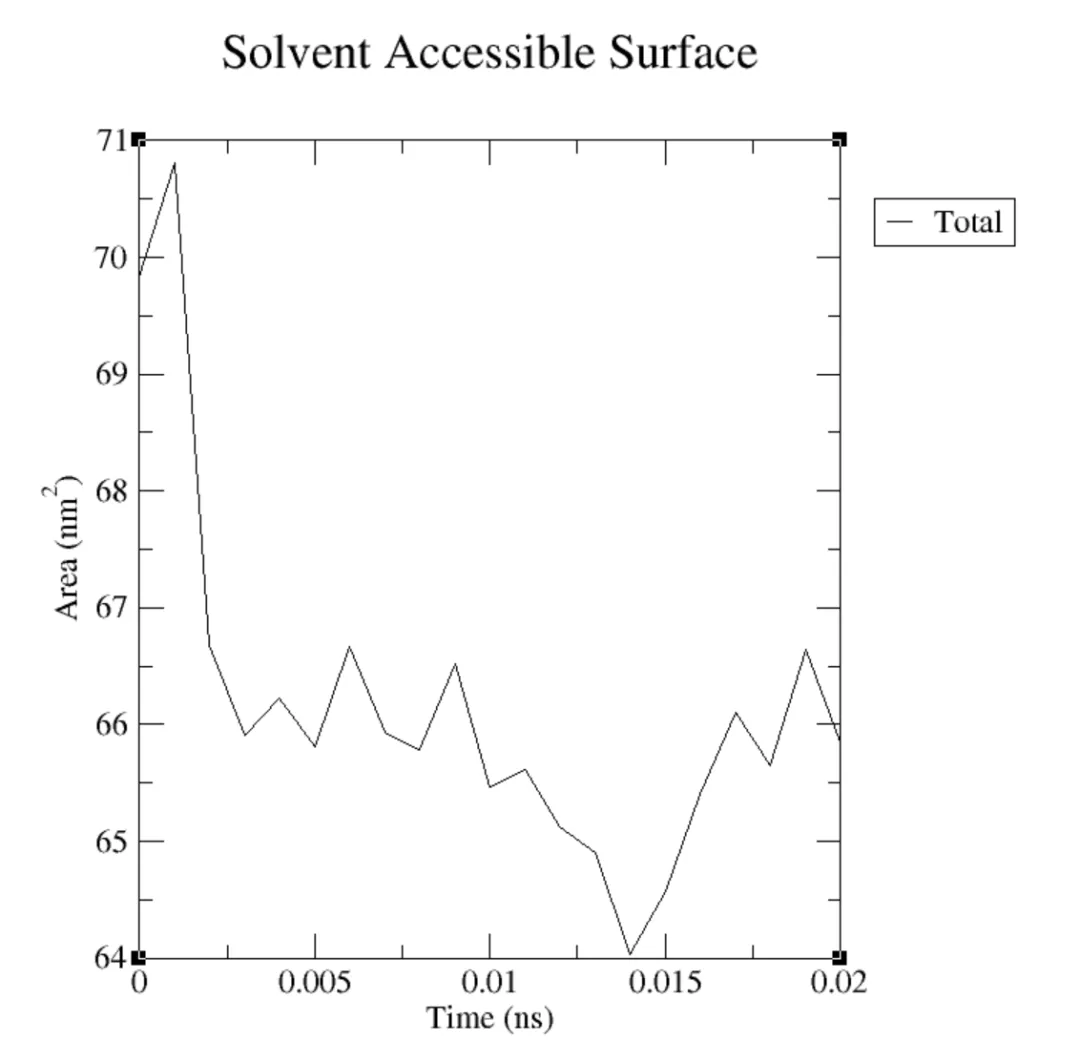
⑦SASA 
⑧自由能形貌图 
具体作图可以看我另一文章以及相关视频2d 3d FEL自由能形貌图 另外FEL需要一个xpm2all.bsh脚本 可以自行去李继存老师github下载 
已经在123老群的小伙伴无需重复进去,无法进入的话下方加我拉群 



推荐课程 

#导出去除水的轨迹以及蛋白文件echo 'non-water' | gmx trjconv -f md.xtc -s md.tpr -o md_nowater.xtcecho 'non-water' | gmx trjconv -f md.gro -s md.tpr -o md_nowater.gro



#查看能量温度压力密度变化等echo -e "Potential\nTemperature\nPressure\nDensity\n" | gmx energy -f md.edrecho -e "Protein\nSystem" | gmx trjconv -s md.tpr -f md.xtc -o md_noPBC.xtc -pbc mol -center




#RMSDecho -e 'backbone \n backbone' | gmx rms -s md.tpr -f md.xtc -o rmsd.xvg -tu ns



#RMSFecho 'protein' | gmx rmsf -f md.xtc -s md.tpr -o rmsf.xvg -res



#RG 径向函数echo -e 'Protein' | gmx gyrate -s md.tpr -f md.xtc -o rg.xvg -tu ns


#second structionecho -e "Protein" | gmx dssp -f md.xtc -s md.tpr -num sec_str.xvg

xmgrace -nxy sec_str.xvg
#SASA xmgrace -nxy area.xvgecho "protein" | gmx sasa -f md.xtc -s md.tpr -o sasa.xvg -tu ns


# 步骤1: 协方差分析echo -e 'C-alpha \n C-alpha \n' | gmx covar -s md.tpr -f md_noPBC.xtc -o eigenvalues.xvg -v eigenvectors.trr -xpma covapic.xpm# 步骤2: 计算第一主成分投影echo -e 'C-alpha \n C-alpha \n' | gmx anaeig -s md.tpr -f md_noPBC.xtc -v eigenvectors.trr -first 1 -last 1 -proj pc1.xvg# 步骤3: 计算第二主成分投影echo -e 'C-alpha \n C-alpha \n' | gmx anaeig -s md.tpr -f md_noPBC.xtc -v eigenvectors.trr -first 2 -last 2 -proj pc2.xvg# 步骤4: 提取pc2数据并与pc1组合awk '{print $2}' pc2.xvg > pc2_values.txtpaste pc1.xvg pc2_values.txt > pc12_shaminput.xvg# 步骤5: 计算自由能形貌图gmx sham -tsham 300 -nlevels 300 -f pc12_shaminput.xvg -ls pc12_gibbs.xpm -g pc_12.log -lsh pc12_enthalpy.xpm -lss pc12_entropy.xpm# 步骤6: 转换为其他格式(如果xpm2all.bsh存在)if [ -f "xpm2all.bsh" ]; then source xpm2all.bsh pc12_gibbs.xpm; else echo "xpm2all.bsh 文件不存在,跳过转换"; fi

https://github.com/Jerkwin/gmxtools/blob/master/xpm2all/xpm2all.bsh
【蛋白模拟后分析实操】 https://www.bilibili.com/video/BV1ptrkBbEFL/?share_source=copy_web&vd_source=76b3de73577fa486f118a69789f8b532

疑问可联系下方UP微信
视频教程点击左下角“阅读原文”跳转B站







本文来自网友投稿或网络内容,如有侵犯您的权益请联系我们删除,联系邮箱:wyl860211@qq.com 。
