大脑图谱到网络模板的映射(含映射文件和代码)
- 2026-02-05 00:38:47
研究背景
大脑图谱(brain atlas) 为脑影像分析提供标准化空间分区,用于提取兴趣区(ROI)层面的结构或功能指标。但仅做区域分析难以体现大脑的系统级功能组织。功能网络从区域协同活动出发刻画大尺度功能结构。
将图谱映射到功能网络,可在保留原有分区的前提下引入网络信息,使 ROI 结果能在功能系统框架中整合与解释,连接区域分析与网络分析。
茗创科技可提供 “从大脑图谱(ROI)到功能网络(如 Yeo 7/17-network atlas)”的标准化映射服务:
在统一标准空间中计算 ROI 与网络模板的重叠归属,输出可复现的映射结果(映射表、网络标签文件、体素数量)以及完整代码/流程(脚本、参数),帮助你在不更换原始图谱的前提下,把 ROI 结果提升到网络层面解释与统计。
详细内容请扫描文章底部二维码咨询
大脑图谱与功能网络的层级差异
大脑图谱:ROI 划分的基础
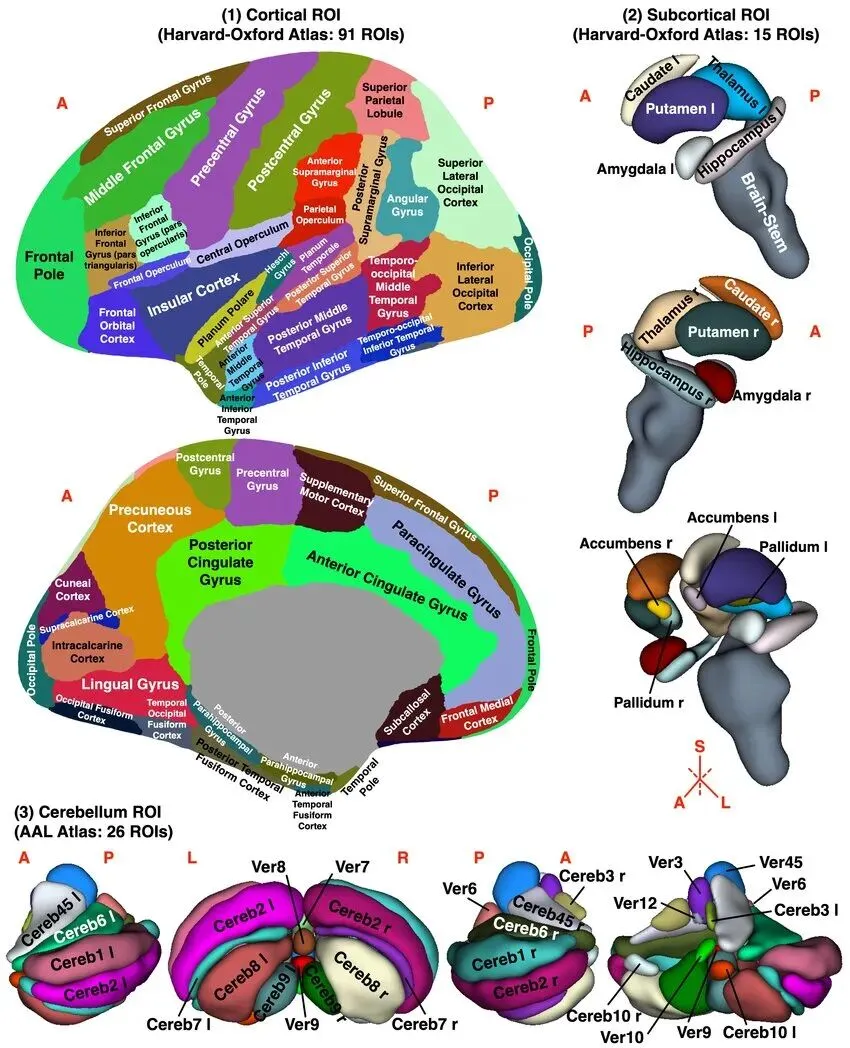
在脑影像分析中,大脑图谱通常用于对全脑进行ROI划分,每个 ROI 对应一个空间上连续的体素集合,用于提取 ROI 层面的影像学指标。
常见的图谱包括基于解剖结构划分的 AAL 和 Harvard–Oxford(HOA)图谱、基于皮层细胞结构的 Brodmann 分区,以及基于功能节点定义的 Power 图谱等。
这类图谱为 ROI 分析提供清晰、可操作的空间单位,便于在不同被试和数据集之间进行一致的指标提取与比较。
功能网络:系统层面的组织框架
与此不同,大脑网络更关注多个脑区之间在功能或连接层面的协同关系,强调大脑活动在系统层面的组织方式,通常以网络或功能系统的形式进行描述。
例如,基于静息态功能连接划分的网络模板(Yeo 7/17)中,常将大脑功能组织概括为若干大尺度网络,如默认模式网络(DMN)、执行控制网络(FPN)、感觉运动网络(SMN) 等。
因此,图谱与网络在分析层级和侧重点上存在差异:前者侧重空间分区,后者侧重区域间关系及其整体组织结构。
大脑图谱到功能网络的映射方法
为什么需要图谱到网络的映射
在实际分析中,为了不改变原有的脑区划分,可以将ROI(来自解剖图谱或结构图谱)与已知的功能网络进行映射,从而建立独立脑区与网络之间的对应关系。这样不仅可以关注脑区的结构信息,还能进一步理解这些脑区在系统层面的功能归属,例如它们更可能属于 DMN、FPN 或 SMN,从而辅助解释脑区在不同认知功能中的角色。
映射思路:标准空间中的重叠归属
具体而言,该方法通常在统一的标准空间中进行:对图谱中的每一个 ROI,计算其与网络模板中各网络区域的空间重叠程度,并将其归属至重叠程度最大的网络单元。
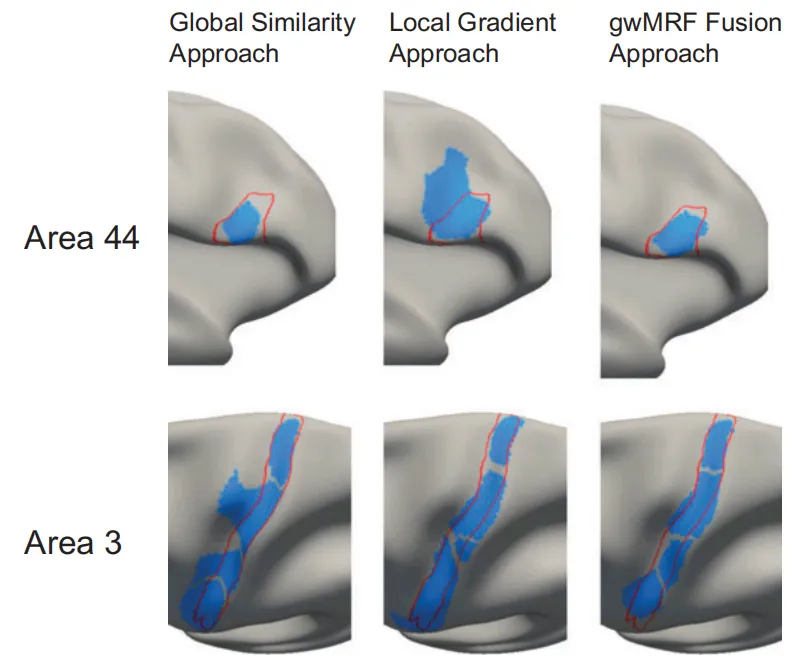
通过这种方式,每个图谱 ROI 都可以被关联到一个主要参与的功能网络,同时保留其原有的空间形态和编号体系。
映射后的用途:从 ROI 结果到网络层面解释
借助这种映射关系,原本基于单个 ROI 的分析结果可以在网络层面进行整理和归纳,例如将属于同一功能网络的多个 ROI 指标进行汇总,或在不同网络之间进行比较。这样能够把零散的区域结果放到更大的功能框架中理解,为从区域层面的描述过渡到系统层面的分析提供自然衔接,同时也有助于在使用不同图谱或分析尺度时保持结果一致性。
功能网络和大脑图谱
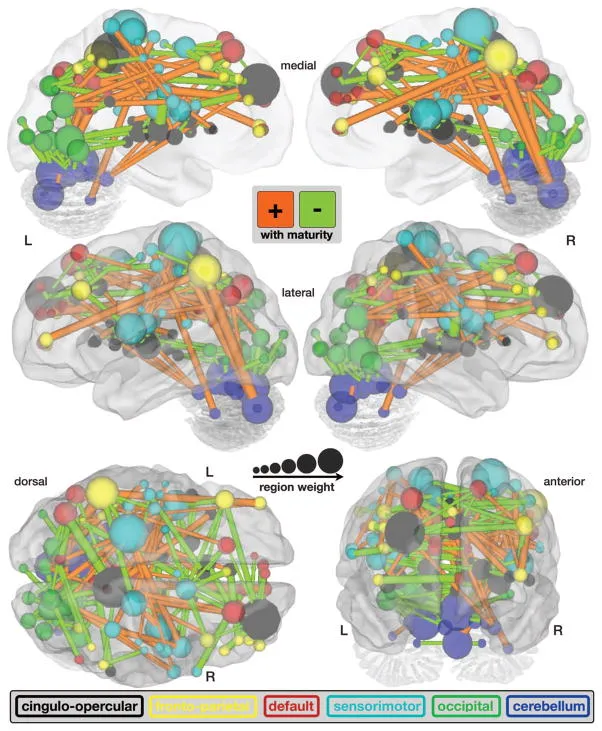
网络模版:Yeo 7 networks
Yeo Atlas(也常称 Yeo 7/17-network atlas)是基于静息态功能磁共振(resting state fMRI)构建的人脑大尺度功能网络图谱,由 B. T. Thomas Yeo 团队在 2011 年发表。它通过对 1000 名受试者全脑静息态功能联结进行聚类,将大脑皮层划分为 7 个或 17 个静息态网络(resting state networks, RSNs)。
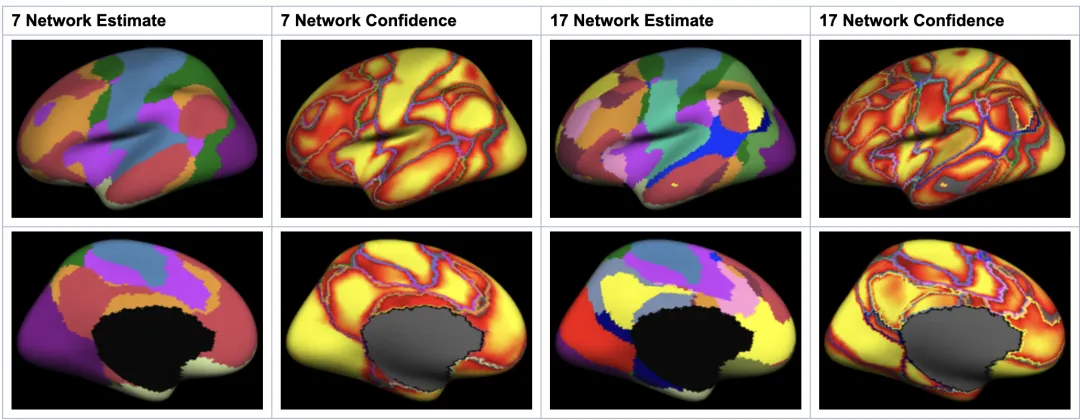
本次网络框架以 Yeo 2011 的 7-network 皮层功能网络划分为主(也常称 Yeo 7/17-network atlas)。
Yeo 7 的常用网络命名如下(不同文献可能会有小幅命名差异):
• Visual(视觉)• Somatomotor(躯体运动)• Dorsal Attention(背侧注意)• Ventral Attention / Salience(腹侧注意/显著性)• Limbic(边缘系统)• Frontoparietal / Executive Control(额顶/执行控制)• Default Mode(默认模式)
大脑图谱
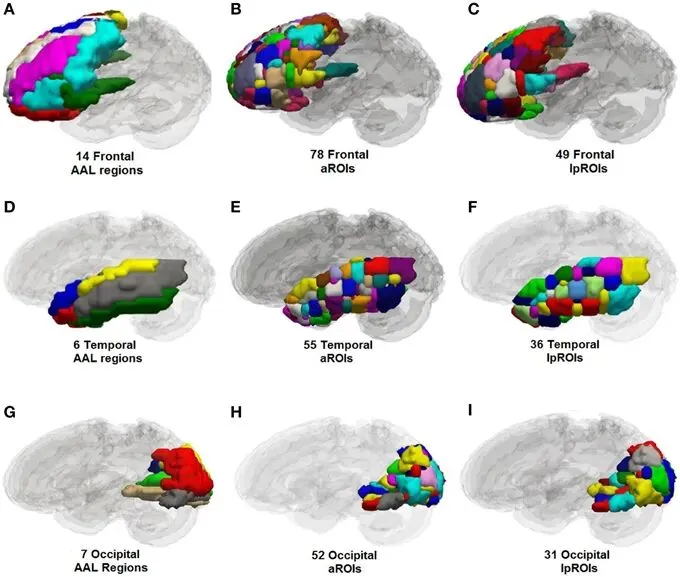
AAL 图谱(Automated Anatomical Labeling)
AAL 属于解剖学图谱,基于标准化到 MNI 空间的结构像,把大脑按解剖边界自动分区并给出标签,便于在 ROI 水平提取信号与做连接分析。常见用法是把体素信号汇总到每个脑区,再进一步计算脑区间相关矩阵。AAL 在工具箱/实现层面有不同版本与标签表,具体 ROI 数量会随版本与是否合并/拆分小脑而变化。
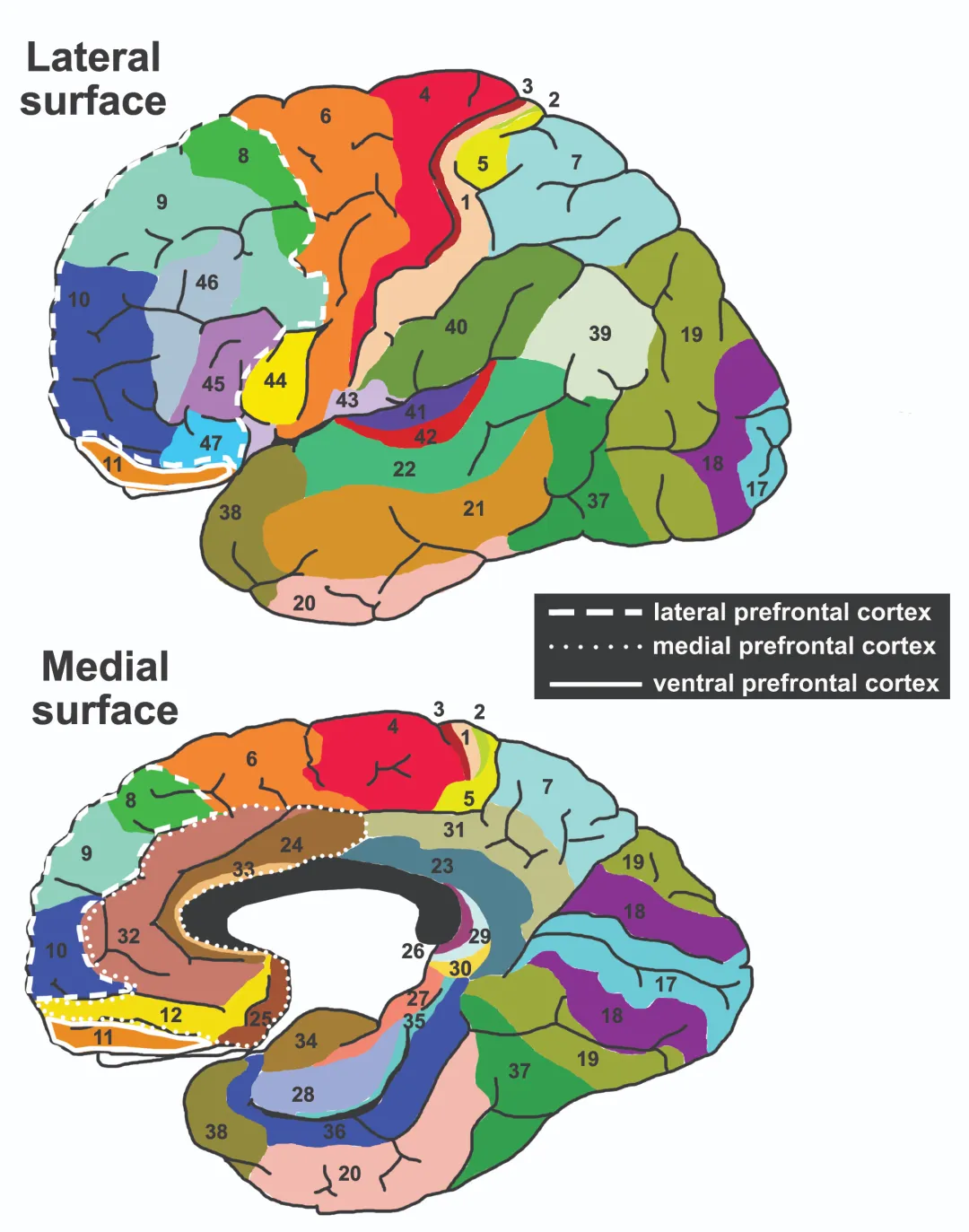
Brodmann Atlas(布罗德曼分区)
Brodmann 分区是经典的皮层细胞结构(cytoarchitecture)分区体系,核心价值在于“以皮层微观结构差异来定义区域”,在解释功能定位、与任务激活/电生理结果对照时很常用。它更像是“皮层分区语言”,常被拿来做结果汇报的参照系,而不是为了精细的全脑功能网络划分。
Harvard–Oxford(HOA)Atlas
HOA 是一套概率型结构分区图谱,覆盖皮层与皮下结构;很多实现来自 FSL 的打包版本,在设定不同概率阈值、是否把左右半球拆分等条件下,最终可用的 ROI 数量会不同。因此“多少个脑区”常常是“某一具体实现的产物”,而不是 HOA 的唯一固定常数。
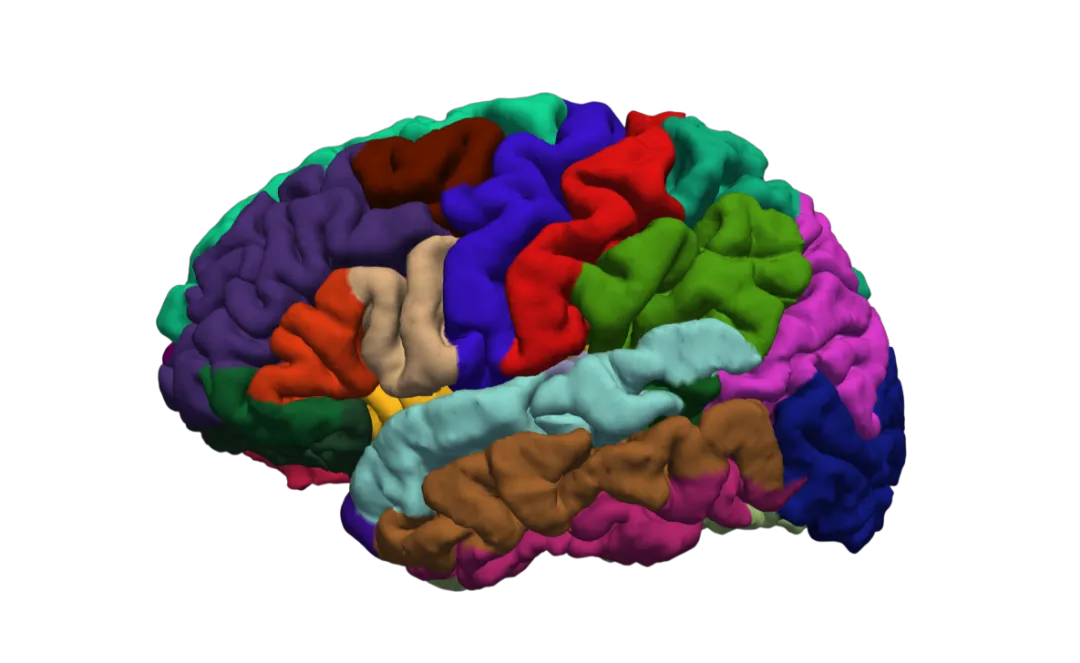
Desikan Atlas(常指 Desikan–Killiany)
Desikan–Killiany 是基于脑沟脑回地标的皮层分区体系,在结构像分析(皮层厚度、表面积等)与表面空间(surface-based)处理中非常常见。很多文献使用“每半球 34 个皮层区,总计约 68 个”的 DK 皮层分区表述;不同项目也会加上额外结构或做轻微变体,从而出现 70 等近邻数字。
Dosenbach Atlas(常见为 Dosenbach-160)
Dosenbach 2010 常被称为“坐标点/节点式”的功能图谱:用一组预定义 ROI 坐标(通常以球形 ROI 表示)覆盖皮层与小脑,并把这些 ROI 归入若干认知/功能网络,常用于功能连接与图论指标计算,优势是“网络标签相对明确、直接对接图论分析”。
AICHA Atlas(384 区)
AICHA 是基于静息态功能连接构建的功能图谱,强调左右半球同源区(homotopic)配对:总计 384 个 ROI(192 对同源区),常用于需要左右可比性或对称性假设的功能连接分析。
Schaefer Atlas
(多尺度分区:200 / 300 / 400 / 1000 个脑区,常用于图论分析)
Schaefer 2018 是非常常用的皮层功能分区方案,提供多尺度 ROI 数(100–1000 都可见),并可附带 7 网络或 17 网络注释(常与 Yeo 网络体系对齐),非常适合做图论分析里的“分辨率敏感性检验”(同一套方法在 200/400/1000 ROI 下是否稳健)。
Power Atlas(264 节点)
Power 2011 提供了一套 264 个功能节点(坐标点)集合,来自功能连接与元分析证据,常用于全脑功能连接与网络分析,是“节点式图谱”的经典代表之一。
Fair Atlas(34 节点)
Fair 等人的早期网络图论工作中使用过较小规模的节点集合(例如 34 节点)来演示发展期网络性质与小世界特征。这里的关键点是“节点数较少、偏演示与方法论情境”,在现代全脑精细连接研究中通常会与更高分辨率分区互为补充。
HCP altas(360区)
HCP360(或 HCP-MMP1.0)是一套基于 Human Connectome Project 年轻成人数据建立的皮层分区图谱,把左右半球各划分为 180 个皮层区(合计 360)。分区边界不是靠单一指标“拍脑袋”,而是综合了皮层髓鞘含量、皮层厚度、任务 fMRI、静息态功能连接与拓扑等多模态证据来确定,所以常被当作“高分辨率、解释性相对更强”的皮层 ROI 方案,用于功能连接、结构连接、图论分析等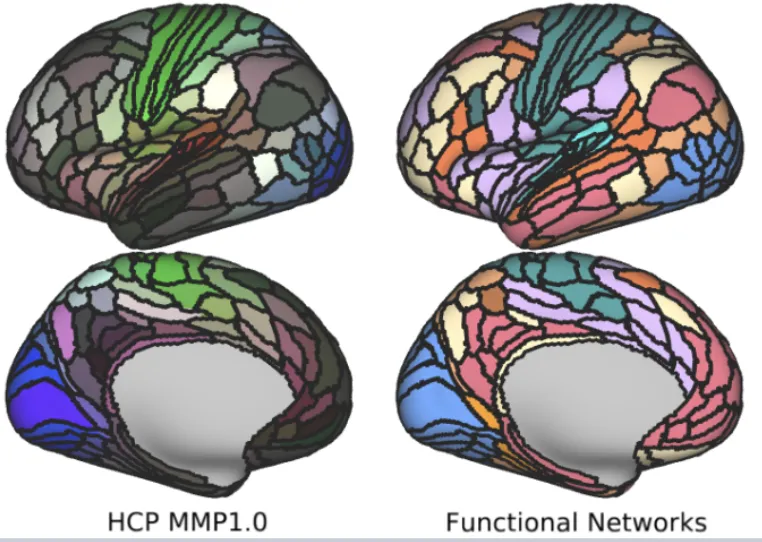
资源与复现
已整理的图谱文件(NIfTI/label table)、网络模板(Yeo 7/17)、以及完整映射代码与运行说明(含环境依赖与版本记录),可扫下方二维码添加企业微信获取
图谱整理参考 neuroparc 与各图谱官方实现;如需与 FreeSurfer/FSL/SPM 工作流对接,可提供对应读入与空间对齐脚本。
扫码获取资源
👇👇👇


图谱和核心代码来源:
工具包
https://github.com/neurodata/neuroparc/tree/master
https://www.freesurfer.net/fswiki/CorticalParcellation_Yeo2011
参考文献:
Brodmann, K. (1909). Vergleichende Lokalisationslehre der Grosshirnrinde in ihren Prinzipien dargestellt auf Grund des Zellenbaues. Johann Ambrosius Barth.
Dosenbach, N. U. F., Nardos, B., Cohen, A. L., Fair, D. A., Power, J. D., Church, J. A., Nelson, S. M., Wig, G. S., Vogel, A. C., Lessov-Schlaggar, C. N., Barnes, K. A., Dubis, J. W., Feczko, E., Coalson, R. S., Pruett, J. R., Jr., Barch, D. M., Petersen, S. E., & Schlaggar, B. L. (2010). Prediction of individual brain maturity using fMRI. Science, 329(5997), 1358–1361.
Fair, D. A., Cohen, A. L., Power, J. D., Dosenbach, N. U. F., Church, J. A., Miezin, F. M., Schlaggar, B. L., & Petersen, S. E. (2009). Functional brain networks develop from a “local to distributed” organization. PLoS Computational Biology, 5(5), e1000381.
FMRIB Software Library. (n.d.). Harvard-Oxford cortical and subcortical structural atlases [Data set]. University of Oxford.
Glasser, M. F., Coalson, T. S., Robinson, E. C., Hacker, C. D., Harwell, J., Yacoub, E., Ugurbil, K., Andersson, J., Beckmann, C. F., Jenkinson, M., Smith, S. M., & Van Essen, D. C. (2016). A multi-modal parcellation of human cerebral cortex. Nature, 536(7615), 171–178.
Jenkinson, M., Beckmann, C. F., Behrens, T. E. J., Woolrich, M. W., & Smith, S. M. (2012). FSL. NeuroImage, 62(2), 782–790.
Joliot, M., Jobard, G., Naveau, M., Delcroix, N., Petit, L., Zago, L., Crivello, F., Mellet, E., Mazoyer, B., & Tzourio-Mazoyer, N. (2015). AICHA: An atlas of intrinsic connectivity of homotopic areas. Journal of Neuroscience Methods, 254, 46–59.
Power, J. D., Cohen, A. L., Nelson, S. M., Wig, G. S., Barnes, K. A., Church, J. A., Vogel, A. C., Laumann, T. O., Miezin, F. M., Schlaggar, B. L., & Petersen, S. E. (2011). Functional network organization of the human brain. Neuron, 72(4), 665–678.
Schaefer, A., Kong, R., Gordon, E. M., Laumann, T. O., Zuo, X.-N., Holmes, A. J., Eickhoff, S. B., & Yeo, B. T. T. (2018). Local-global parcellation of the human cerebral cortex from intrinsic functional connectivity MRI. Cerebral Cortex, 28(9), 3095–3114.
Tzourio-Mazoyer, N., Landeau, B., Papathanassiou, D., Crivello, F., Etard, O., Delcroix, N., Mazoyer, B., & Joliot, M. (2002). Automated anatomical labeling of activations in SPM using a macroscopic anatomical parcellation of the MNI MRI single-subject brain. NeuroImage, 15(1), 273–289.
Yeo, B. T. T., Krienen, F. M., Sepulcre, J., Sabuncu, M. R., Lashkari, D., Hollinshead, M., Roffman, J. L., Smoller, J. W., Zöllei, L., Polimeni, J. R., Fischl, B., Liu, H., & Buckner, R. L. (2011). The organization of the human cerebral cortex estimated by intrinsic functional connectivity. Journal of Neurophysiology, 106(3), 1125–1165.
Glasser, M. F., Coalson, T. S., Robinson, E. C., Hacker, C. D., Harwell, J., Yacoub, E., Ugurbil, K., Andersson, J., Beckmann, C. F., Jenkinson, M., Smith, S. M., & Van Essen, D. C. (2016). A multi-modal parcellation of human cerebral cortex. Nature, 536(7615), 171–178.
脑电数据分析启航班(训练营:2026.3.27~4.25)
核磁培训班
磁共振弥散张量成像(DTI)数据处理班(训练营:2026.2.12~2.20,新增DTI-ALPS)
磁共振弥散张量成像(DTI)数据处理进阶班(训练营:2026.2.23~3.6)
热门课程
全学科!顶级DeepSeek学术指令!一周完成高质量论文(直播:2026.1.24~1.25)
DeepSeek智能辅助科研标书写作实操指南课程(直播:2026.1.24~1.25)
数据处理业务
会员系列
相关说明
本文内容仅供参考,一切内容以参考文献中的原文为准。
非常欢迎大家留言、转载、收藏或分享~
本文来自微信公众号“茗创科技”。如需转载,请在“茗创科技”后台回复“转载”,并附上所需转载的文章标题以及您的ID。
茗创科技
MC_Brain
觉得有帮助,欢迎转发收藏或者点个在看哦~
听说点在看的人SCI接收率都提升了18%呢!

