AFNI、FSL和ANTs做fMRI数据预处理的文章,小伙伴们都挺感兴趣,这次文章在上次公众号文章的基础上对数据预处理进行详细的解释,并附上相关的命令。一、数据格式转换(DICOM → NIfTI)
MRI扫描仪输出的是 DICOM 格式文件。为了后续分析,需要转换为 NIfTI(.nii或.nii.gz)格式(MRIcron工具包)。
dcm2niix -o ./output_dir -f sub01_func ./DICOM_func
二、去除前若干时间点
fMRI扫描开始时,磁化信号尚未稳定,前几个时间点信号偏低或不稳定,通常去除前5–10 个TR/时间点(FSL工具包)。
fslroi func func_trimmed 10 -1
三、时间校正(Slice Timing Correction)
MRI的每个体积(volume)由多个切片(slice)顺序采集,各切片采集时间不同。该步骤将所有切片的信号时间点校正到同一时间基准,以保证时间序列同步(AFNI工具包)。
3dTshift -TR 2.0 -tpattern alt+z -prefix func_stc func_trimmed.nii
四、头动校正(Motion Correction)
被试在扫描期间会发生轻微头动,导致体素位置变化。通过配准算法将每个时间点的体积对齐到参考体积。头动参数(6个:3平移 + 3旋转),后续可作为共变量回归项(AFNI工具包)。
3dvolreg \
-prefix func_mc.nii.gz \
-base 0 \
-1Dfile motion.1D \
-1Dmatrix_save mat.aff12.1D \
-Fourier \
func_stc.nii.gz
五、配准(Coregistration)
将功能图像(低分辨率)与结构图像(高分辨率T1)对齐。保证功能信号的定位在解剖结构上准确(ANTs工具包)。
antsRegistrationSyNQuick.sh \
-d 3 \
-f T1_brain.nii.gz \
-m mean_func.nii.gz \
-o coreg_
六、标准化(Normalization)
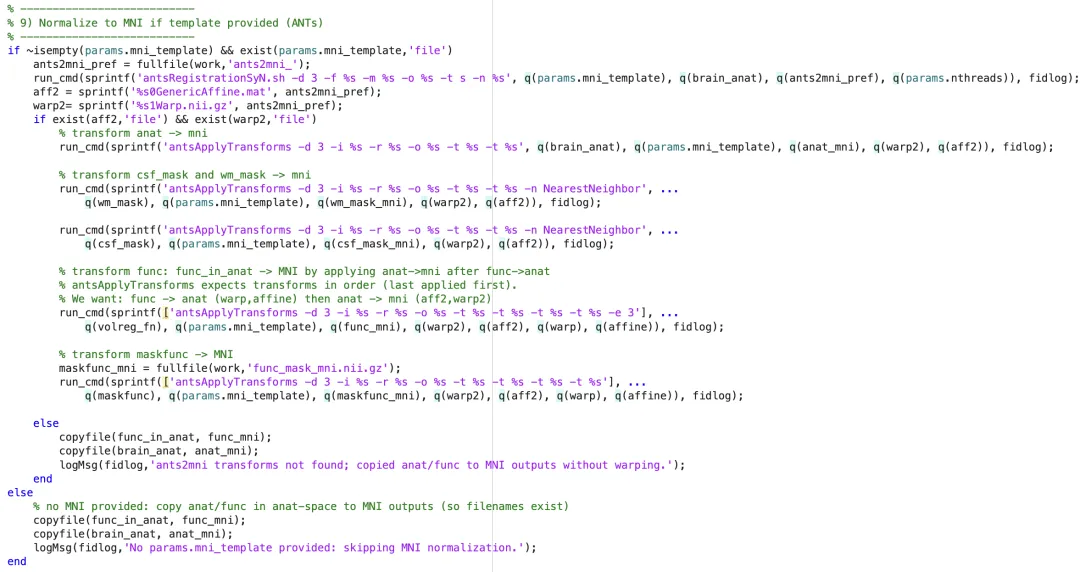
标准化(Normalization)是将个体的结构或功能图像非线性映射到标准空间(如 MNI152),以便进行群体水平的统计分析和结果可视化 (ANTs工具包)。
(1)结构像标准化(T1 → MNI)
antsRegistrationSyN.sh \
-d 3 \
-f MNI152_T1_1mm_brain.nii.gz \
-m T1_brain.nii.gz \
-o norm_
(2) 功能图像标准化(fMRI → MNI)
antsApplyTransforms \
-d 3 \
-e 3 \
-i func_coreg.nii.gz \
-r MNI152_T1_2mm_brain.nii.gz \
-o func_MNI.nii.gz \
-t norm_1Warp.nii.gz \
-t norm_0GenericAffine.mat
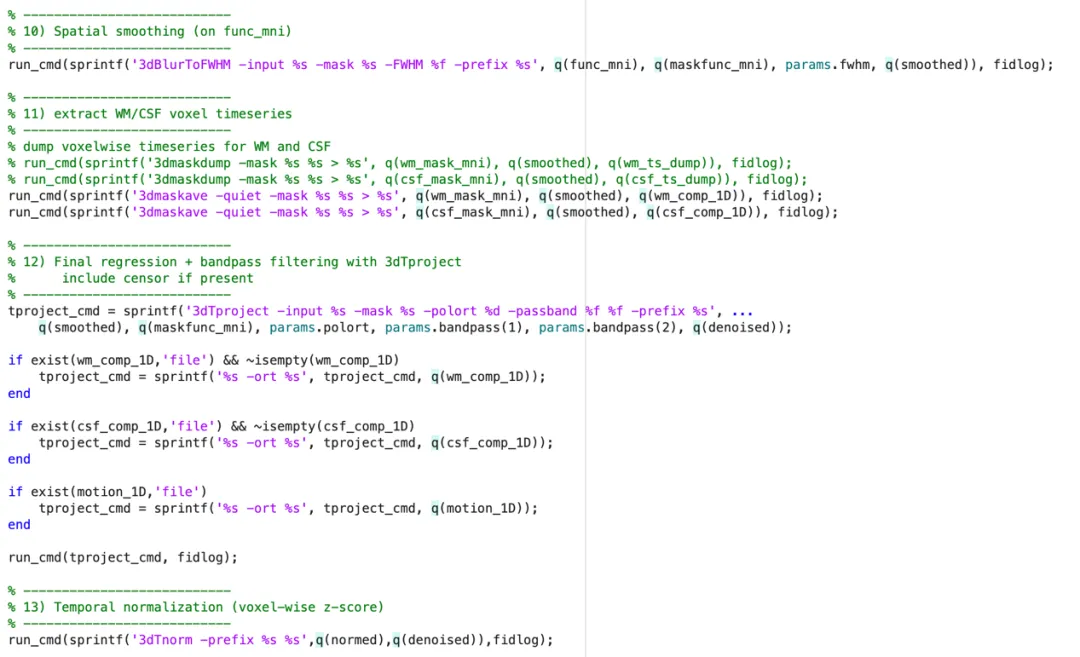
七、空间平滑(Spatial Smoothing)
空间平滑(Spatial Smoothing)是在空间维度上对 BOLD 图像进行高斯滤波,提高信噪比(减少高频噪声),满足统计分析的随机场理论假设(如SPM的随机场推断)弥合不同受试者的解剖差异,改善空间连续性,使统计信号更稳健(AFNI工具包)。
3dBlurToFWHM \
-input func_MNI.nii.gz \
-FWHM 6.0 \
-automask \
-prefix func_MNI_smooth.nii.gz \
-blurmaster func_MNI.nii.gz
八、共变量回归 + 时间滤波
头动影响(6个运动参数)、生理噪声(白质WM、脑脊液CSF信号)、低频漂移 / 高频噪声(AFNI工具包)。
3dTproject \
-input func_MNI_smooth.nii.gz \
-mask brain_mask.nii.gz \
-ort motion.1D \
-ort WM_CSF.1D \
-bandpass 0.01 0.1 \
-blur 0 \
-polort 2 \
-prefix func_MNI_clean.nii.gz
九、最后附上Matlab整合代码
(1)生成文件命名

(2)预处理代码



最后,有数据处理方面的疑问和讨论其他科研问题可以后台私信我呦。