在生物学研究中,如何利用实验室培养的干细胞精准模拟人类早期胚胎发育,一直是生命科学领域的一个核心议题。由于人类早期胚胎样本获取极其受限,研究人员一直在寻找能够稳定模拟胚胎基因组激活(EGA)的体外模型。
2026年1月,赫尔辛基大学的Sanna Vuoristo团队在《Cell Reports》杂志发表研究,通过整合转录组分析揭示了与人类8细胞期样细胞(8CLCs)相关的代谢重塑和基因表达网络。今天我们就来拆解一下这篇文章:Integrated transcriptomic analysis reveals metabolic remodeling and gene expression networks related to human 8-cell-stage embryo-like cells。

研究概述
人类早期发育研究面临样本稀缺和细胞数量少的挑战。8细胞期样细胞(8CLCs)的出现为理解人类胚胎基因组激活提供了机会,但不同诱导系统产生的细胞在多大程度上反映了真实胚胎发育尚不明确。本研究整合了多个诱导系统的单细胞转录组数据,通过比对真实胚胎参考系,识别了8CLCs中的关键基因模块、转座子表达及生物学过程。研究确定了成熟态与中间态8CLCs的区别,并揭示了代谢重塑、RNA剪接和核糖体生物合成在其中的作用。

实验设计
研究团队收集了包括自发产生、DUX4诱导及化学诱导在内的多种8CLC诱导系统的单细胞转录组原始数据。这些数据被统一比对至GRCh38人类参考基因组,并与已发表的人类卵子及胚胎数据进行整合分析。实验通过生物信息学手段对细胞进行聚类、细胞周期评分、基因共表达网络(GRN)分析以及转座子(TE)表达量化。此外,研究人员还利用流式细胞术验证了细胞周期分布,并使用Seahorse XF分析仪检测了DUX4诱导后的线粒体呼吸和糖酵解活性。
研究结果
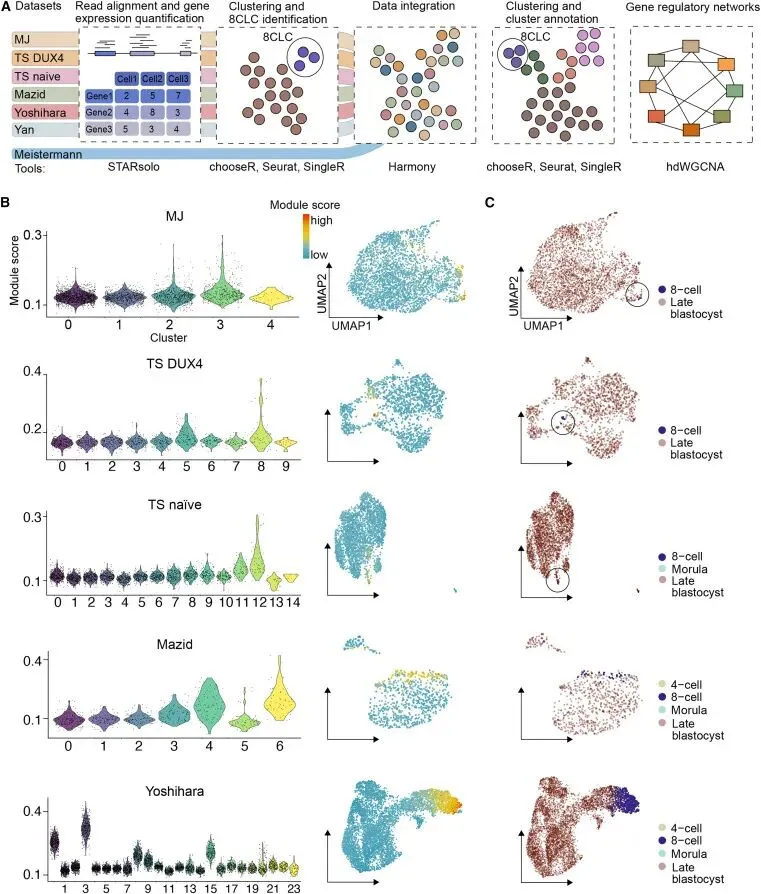 图1:单细胞转录组分析确定了不同数据集中的8CLC簇,并揭示了成熟态与中间态细胞共存的现象。
图1:单细胞转录组分析确定了不同数据集中的8CLC簇,并揭示了成熟态与中间态细胞共存的现象。
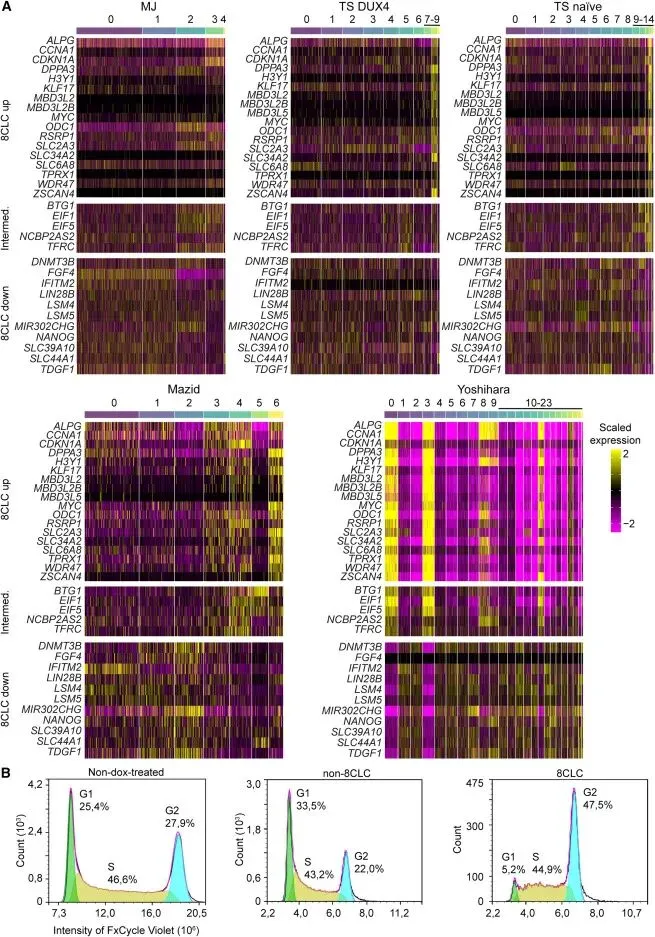
图2:8CLC簇共享了TPR X1、ZSCAN4等已知胚胎标志物,且这些细胞普遍富集在细胞周期的G2期。
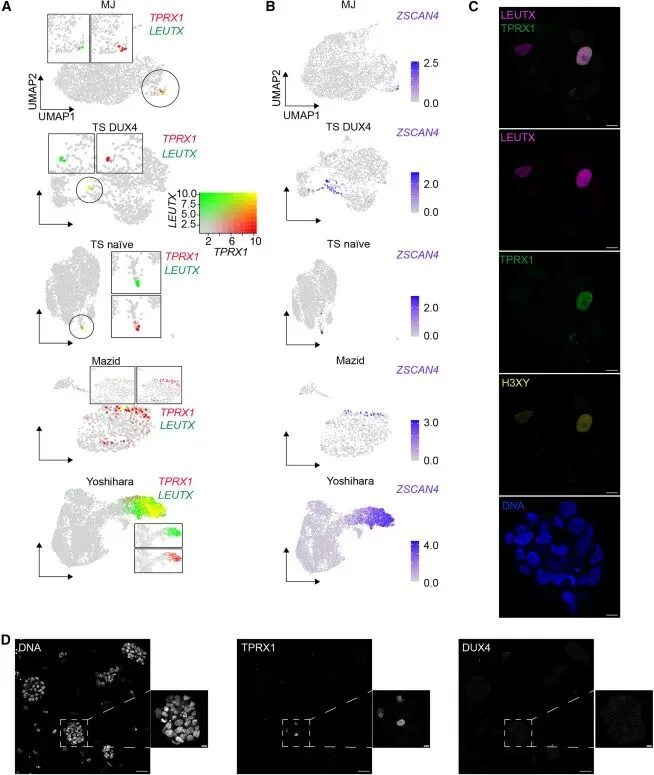
图3:在蛋白水平上证实了TPRX1、H3X/Y和LEUTX在8CLCs中的共表达。
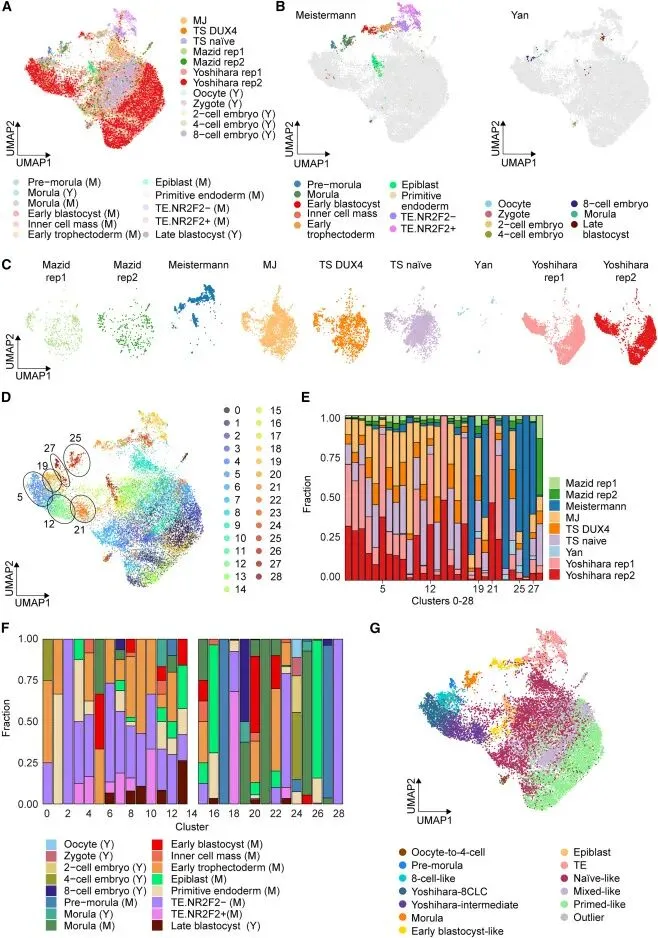 图4:多数据集整合分析显示,Yoshihara和Mazid的数据集在转录谱上最接近真实的人类8细胞期胚胎。
图4:多数据集整合分析显示,Yoshihara和Mazid的数据集在转录谱上最接近真实的人类8细胞期胚胎。
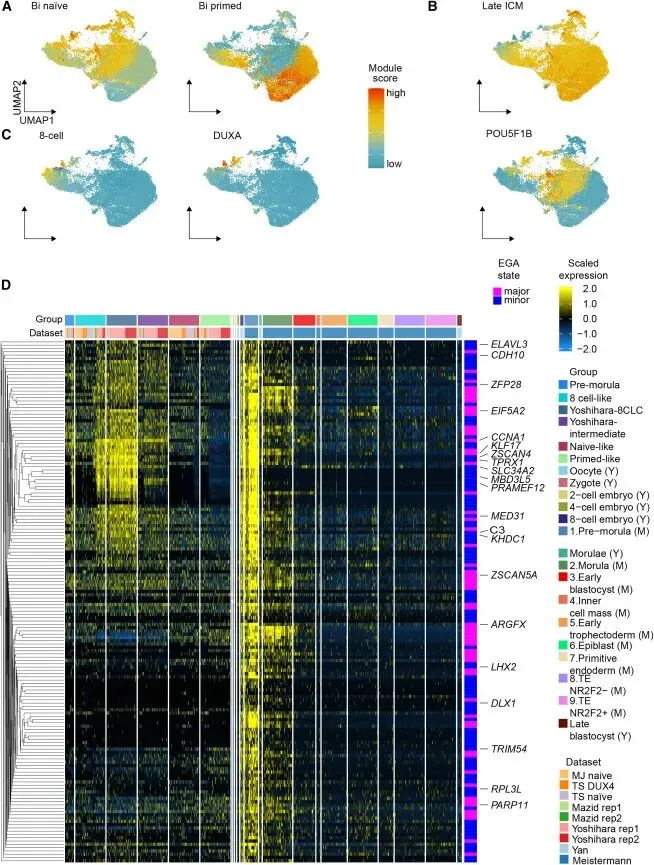
图5:中间态8CLCs仍保留部分干细胞特性,而成熟态8CLCs则实现了EGA相关基因的全面激活和干细胞标记物的下调。
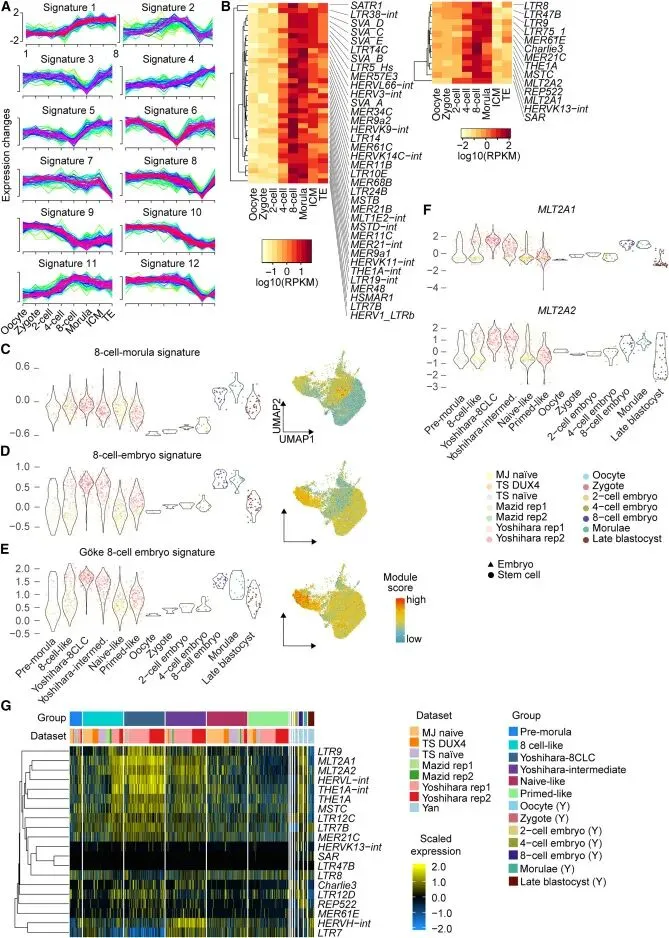 图6:转座子表达特征分析进一步区分了中间态和成熟态,证实成熟态8CLCs能更完整地复现胚胎期的转座子激活图谱。
图6:转座子表达特征分析进一步区分了中间态和成熟态,证实成熟态8CLCs能更完整地复现胚胎期的转座子激活图谱。
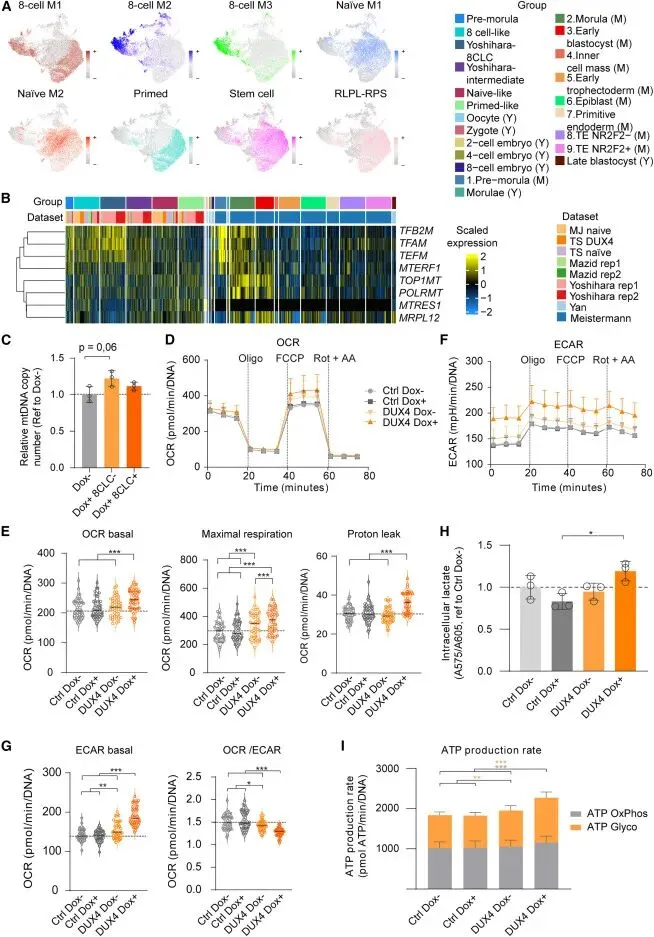
图7:基因共表达网络与功能实验表明,8CLCs的产生伴随着能量代谢模式的转变,表现为由DUX4驱动的糖酵解增强和线粒体呼吸改变。
数据分析
生信分析
本研究涉及的组学技术包括单细胞转录组测序(scRNA-seq)、单细胞多组学中的RNA部分(single-cell Multiome RNA)和转座元件表达分析(scTE)。
分析流程
- 1. 涉及组学技术:单细胞RNA测序(scRNA-seq)、转座元件表达分析(scTE)。
- • 数据预处理:将原始测序数据用STARsolo比对到GRCh38人类参考基因组,进行质量控制,过滤低质量细胞和基因。
- • 聚类分析:使用Seurat进行标准化、降维和分群,通过chooseR优化聚类分辨率,结合UMAP进行可视化。
- • 细胞注释:利用SingleR算法,以人类胚胎和干细胞数据为参考,对细胞发育阶段和身份进行注释。
- • 差异表达与功能富集:识别8CLC集群的差异表达基因,通过GO分析进行功能富集,明确核心生物学过程。
- • 转座元件分析:使用scTE量化TE表达,结合Mfuzz进行模糊聚类以识别胚胎阶段特异性TE表达轨迹。
- • 基因调控网络构建:通过hdWGCNA来构建共表达网络,识别驱动8CLC状态的核心枢纽基因和调控模块。
- • 数据整合:利用Harmony整合多个数据集,消除批次效应,进行跨数据集比较分析。
统计分析
分类变量如细胞周期分布采用卡方检验。差异表达分析使用Seurat的Wilcoxon秩和检验,并采用Benjamini-Hochberg方法进行多重假设检验校正。代谢实验数据(如OCR、ECAR、乳酸水平等)则采用单因素或混合效应方差分析(ANOVA),并辅以Tukey's HSD事后检验。文中所有统计显著性阈值设定为p < 0.05。
总结
研究意义
本研究明确了不同来源8CLCs的异质性,区分了成熟与中间态细胞特征,揭示了代谢重编程、转座元件激活和基因调控网络在8CLCs形成中的关键作用。其发现为优化8CLC体外模型提供了分子依据,助力深入解析人类胚胎基因组激活机制,对生殖医学和发育生物学研究具有重要参考价值。
文章复现
这篇文章的原始数据和生信分析代码都公开了,非常全面。
原始数据及仓库地址如下:
- • 8CLC相关scRNA-seq数据:SRA(SRR14853531、SRR16975081、SRR18215263、SRR18215264、SRR18215265、SRR18215266)、ArrayExpress(E-MTAB-10581)、CNGB Nucleotide Sequence Archive(CNX0278328、CNX0278329)
- • 干细胞scRNA-seq数据:SRA(SRR19353580、SRR19353578)
- • 预处理的人类胚胎scRNA-seq数据:GEO(GSE36552)、Mendeley Data(https://doi.org/10.17632/689p.m.8s7jc.1)
- • 生信分析代码:Zenodo(https://doi.org/10.5281/zenodo.12517517)
推荐阅读
中国银河生信云平台(UseGalaxy.cn)致力于零代码生信分析。平台拥有海量计算资源、3000 多个生信工具和数十条生信流程,并且为用户提供 200G 免费存储空间。进群交流请先加 usegalaxy 为好友。我们还为进阶用户提供高质量培训课程:
RNA-seq数据分析实战 | 2026年第2期,开启你的生信学习之旅